Objetivos
- Compare ponto de mutações e mutações frameshift
- Descrever as diferenças entre missense, absurdo, e em silêncio mutações
- Descrever as diferenças entre o claro e o escuro reparar
- Explicar como diferentes agentes mutagénicos act
- Explique por que o teste de Ames pode ser usado para detectar substâncias cancerígenas
- Análise de sequências de DNA e identificar exemplos de tipos de mutações
uma mutação é Uma hereditários alteração na sequência de DNA de um organismo., O organismo resultante, chamado mutante, pode ter uma mudança reconhecível no fenótipo em comparação com o tipo selvagem, que é o fenótipo mais comumente observado na natureza. Uma mudança na sequência de DNA é conferida ao ARNm através da transcrição, e pode levar a uma sequência alterada de aminoácidos em uma proteína na tradução. Como as proteínas realizam a grande maioria das funções celulares, uma mudança na sequência de aminoácidos em uma proteína pode levar a um fenótipo alterado para a célula e organismo.,
efeitos das mutações na sequência de ADN
Existem vários tipos de mutações que são classificadas de acordo com a forma como a molécula de ADN é alterada. Um tipo, chamado de mutação de ponto, afeta uma única base e mais comumente ocorre quando uma base é substituída ou substituída por outra. As mutações também resultam da adição de uma ou mais bases, conhecidas como inserção, ou da remoção de uma ou mais bases, conhecidas como deleção.que tipo de mutação ocorre quando um gene tem menos dois nucleótidos na sua sequência?,
efeitos das mutações na estrutura e função proteicas
mutações pontuais podem ter uma vasta gama de efeitos na função proteica (Figura 1). Como consequência da degenerescência do código genético, uma mutação pontual resultará normalmente na incorporação do mesmo aminoácido no polipéptido resultante, apesar da mudança de sequência. Esta mudança não teria qualquer efeito sobre a estrutura da proteína, sendo assim chamada de mutação silenciosa. Uma mutação missense resulta na incorporação de um aminoácido diferente no polipéptido resultante., O efeito de uma mutação missense depende de quão quimicamente diferente o novo aminoácido é do aminoácido de tipo selvagem. A localização do aminoácido alterado dentro da proteína também é importante. Por exemplo, se o aminoácido alterado é parte do local ativo da enzima, então o efeito da mutação missense pode ser significativo. Muitas mutações de missense resultam em proteínas que ainda são funcionais, pelo menos em algum grau. Às vezes, os efeitos das mutações de missense podem ser apenas aparentes sob certas condições ambientais; tais mutações de missense são chamadas mutações condicionais., Raramente, uma mutação missense pode ser benéfica. Sob as condições ambientais adequadas, este tipo de mutação pode dar ao organismo que o alberga uma vantagem seletiva. Ainda outro tipo de mutação de ponto, chamada de mutação sem sentido, converte um codon codificando um aminoácido (um codon sentido) em um codon stop (um codon sem sentido). Mutações absurdas resultam na síntese de proteínas que são mais curtas do que o tipo selvagem e tipicamente não funcionais.as supressões e inserções também causam vários efeitos., Uma vez que os codões são trigémeos de nucleótidos, inserções ou deleções em grupos de três nucleótidos podem levar à inserção ou eliminação de um ou mais aminoácidos e não podem causar efeitos significativos na funcionalidade da proteína resultante. No entanto, mutações de frameswift, causadas por inserções ou deleções de um número de nucleotídeos que não são um múltiplo de três são extremamente problemáticas porque uma mudança nos resultados da estrutura de leitura (Figura 1). Como os ribossomas lêem o ARNm nos codões de tripleto, as mutações de transformação podem mudar todos os aminoácidos após o ponto da mutação., O novo quadro de leitura também pode incluir um codon de paragem antes do fim da sequência de codificação. Consequentemente, as proteínas feitas a partir de genes que contêm mutações de mudança de regime são quase sempre não funcionais.
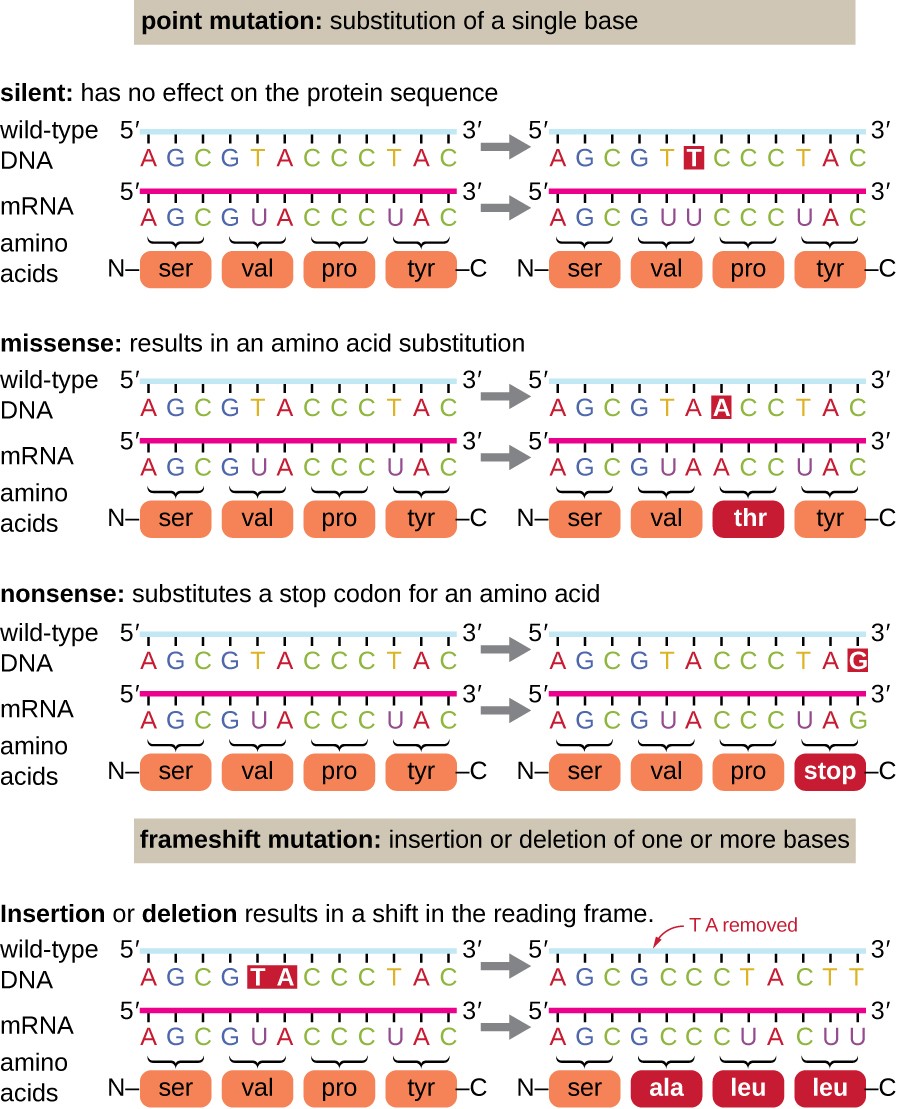
Figura 1. Clique para obter uma imagem maior. As mutações podem levar a alterações na sequência proteica codificada pelo ADN.
pense sobre isso
- Quais são as razões pelas quais uma mudança de nucleótido num gene para uma proteína pode não ter qualquer efeito no fenótipo desse gene?,é possível uma inserção de três nucleótidos juntos após o quinto nucleótido num gene que codifica proteínas para produzir uma proteína que é mais curta do que o normal? Como ou como não?
uma mutação benéfica
desde que o primeiro caso de infecção pelo vírus da Imunodeficiência Humana (VIH) foi notificado em 1981, quase 40 milhões de pessoas morreram de infecção pelo VIH, o vírus que causa a síndrome da Imunodeficiência Adquirida (SIDA)., O vírus destina-se às células T auxiliares que desempenham um papel fundamental na ligação da resposta imunitária inata e adaptativa, infectando e matando as células normalmente envolvidas na resposta do organismo à infecção. Não há cura para a infecção pelo HIV, mas muitos medicamentos foram desenvolvidos para retardar ou bloquear a progressão do vírus. Embora os indivíduos em todo o mundo possam estar infectados, a maior prevalência entre as pessoas de 15-49 anos é na África Subsaariana, onde quase uma pessoa em cada 20 está infectada, representando mais de 70% das infecções em todo o mundo (Figura 2)., Infelizmente, esta é também uma parte do mundo onde as estratégias de prevenção e as drogas para tratar a infecção são as mais carentes.

Figura 2. O HIV é altamente prevalente na África Subsaariana, mas sua prevalência é bastante baixa em algumas outras partes do mundo.
Nos últimos anos, o interesse científico tem sido despertado pela descoberta de alguns indivíduos do norte da Europa, que são resistentes à infecção pelo HIV. Em 1998, o geneticista americano Stephen J., O’Brien do National Institutes of Health (NIH) e colegas publicaram os resultados de sua análise genética de mais de 4.000 indivíduos. Estes indicaram que muitos indivíduos de descendência eurasiana (até 14% em alguns grupos étnicos) têm uma mutação por deleção, chamada CCR5-delta 32, na codificação genética CCR5. CCR5 é um coreceptor encontrado na superfície das células T que é necessário para muitas estirpes do vírus para entrar na célula hospedeira. A mutação leva à produção de um receptor ao qual o VIH não se pode ligar eficazmente e, portanto, bloqueia a entrada viral., As pessoas homozigóticas para esta mutação têm reduzida susceptibilidade à infecção pelo VIH, e aqueles que são heterozigóticos também têm alguma protecção contra a infecção.
não é claro por que as pessoas de ascendência do Norte da Europa, especificamente, carregam esta mutação, mas sua prevalência parece ser mais alta no norte da Europa e diminui constantemente nas populações à medida que se move para o sul. Pesquisas indicam que a mutação tem estado presente desde antes do aparecimento do HIV e pode ter sido selecionada em populações europeias como resultado da exposição à peste ou varíola., Esta mutação pode proteger os indivíduos da peste (causada pela bactéria Yersinia pestis) e varíola (causada pelo vírus da varíola) porque este receptor também pode estar envolvido nestas doenças. A idade desta mutação é uma questão de debate, mas as estimativas sugerem que ela apareceu entre 1875 anos a 225 anos atrás, e pode ter sido espalhada do Norte da Europa através de invasões Viking.esta descoberta excitante levou a novas pistas na pesquisa do HIV, incluindo a busca de drogas para bloquear a ligação do CCR5 ao HIV em indivíduos que não possuem a mutação., Embora testes de DNA para determinar quais indivíduos carregam a mutação CCR5-delta 32 é possível, existem casos documentados de indivíduos homozigóticos para a mutação contraindo HIV. Por esta razão, o teste de DNA para a mutação não é amplamente recomendado pelos funcionários de saúde pública, de modo a não incentivar o comportamento de risco em quem carrega a mutação. No entanto, inibir a ligação do VIH ao CCR5 continua a ser uma estratégia válida para o desenvolvimento de terapias medicamentosas para os infectados pelo VIH.,
causas de mutações
erros no processo de replicação do ADN podem causar a ocorrência de mutações espontâneas. A taxa de erro da DNA polimerase é uma base incorreta por bilhão de pares de base replicados. A exposição a mutagénicos pode causar mutações induzidas, que são vários tipos de agentes químicos ou radiação (Quadro 1). A exposição a um mutagénico pode aumentar a taxa de mutação mais de 1000 vezes. Os mutagénicos são também frequentemente cancerígenos, agentes que causam cancro. No entanto, enquanto quase todos os agentes cancerígenos são mutagénicos, nem todos os agentes mutagénicos são necessariamente cancerígenos.,
| Tabela 1., to AT base pair | Point | |||
|---|---|---|---|---|
| Intercalating agents | ||||
| Acridine orange, ethidium bromide, polycyclic aromatic hydrocarbons | Distorts double helix, creates unusual spacing between nucleotides | Introduces small deletions and insertions | Frameshift | |
| Ionizing radiation | ||||
| X-rays, γ-rays | Forms hydroxyl radicals | Causes single- and double-strand DNA breaks | Repair mechanisms may introduce mutations | |
| X-rays, γ-rays | Modifies bases (e.,g., deaminating C a U) | Converte o GC EM pares de base | Ponto | |
| radiações não ionizantes | ||||
| Ultravioleta | Formas de pirimidina (geralmente timina) dímeros | Faz com que a replicação do ADN erros | Frameshift ou ponto | |
Química Mutagénicas
Vários tipos de química mutagénicas interagem diretamente com o DNA, seja por agir como inibidores nucleosídeos análogos, ou pela modificação de bases de nucleotídeos., As substâncias químicas denominadas análogos nucleósidos são estruturalmente semelhantes às bases nucleótidas normais e podem ser incorporadas no ADN durante a replicação (Figura 3). Estes análogos de base induzem mutações porque muitas vezes têm regras de emparelhamento de base diferentes das bases que substituem. Outros mutagénicos químicos podem modificar bases de ADN normais, resultando em diferentes regras de emparelhamento de bases. Por exemplo, ácido nitroso desamina citosina, convertendo-a em uracil. Uracilo então faz pares com adenina em uma subsequente rodada de replicação, resultando na conversão de um par de base GC para um par de base AT., O ácido nitroso também desaminata a adenina para a hipoxantina, que faz pares de base com citosina em vez de timina, resultando na conversão de um par de base TA para um par de base CG.

Figura 3. Clique para obter uma imagem maior. (a) 2-aminopurine nucleosídeos (2AP) estruturalmente é um nucleosídeo análogo ao de adenina de nucleosídeo, enquanto 5-bromouracil (5BU) é um nucleosídeo análogo a timina nucleosídeos. Pares de base 2AP com C, convertendo um par de base AT para um par de base GC após várias rodadas de replicação., 5BU pares com G, convertendo um par de base AT para um par de base GC após várias rodadas de replicação. (b) ácido nitroso é um tipo diferente de mutagênico químico que modifica bases nucleósidas já existentes como C para produzir U, que pares de base com A. Esta modificação química, como mostrado aqui, resulta na conversão de um par de base CG para um par de base TA.mutagénicos químicos conhecidos como agentes intercalantes funcionam de forma diferente., Estas moléculas deslizam entre as bases azotadas empilhadas da dupla hélice de DNA, distorcendo a molécula e criando espaçamento atípico entre pares de bases nucleotídeos (Figura 4). Como resultado, durante a replicação do DNA, a DNA polimerase pode saltar a replicação de vários nucleótidos (criando uma deleção) ou inserir nucleótidos extras (criando uma inserção). Qualquer resultado pode levar a uma mutação de transformação. Os produtos de combustão, como os hidrocarbonetos aromáticos policíclicos, são agentes intercaladores particularmente perigosos que podem conduzir a cancros provocados por mutações., Os agentes intercalantes brometo de etídio e laranja acridina são comumente usados no laboratório para manchar o DNA para visualização e são mutagênicos potenciais.

Figura 4. Agentes intercalantes, como a acridina, introduzem espaçamento atípico entre pares de bases, resultando em DNA polimerase introduzindo uma exclusão ou uma inserção, levando a uma potencial mutação frameshift.
radiação
exposição a radiações ionizantes ou não ionizantes pode induzir mutações no ADN, embora através de diferentes mecanismos., Uma forte radiação ionizante, como raios-X e raios gama, pode causar quebras de cadeia simples e dupla na espinha dorsal do DNA através da formação de radicais hidroxilo em exposição à radiação (Figura 5). A radiação ionizante também pode modificar bases; por exemplo, a desaminação da citosina ao uracilo, análoga à ação do ácido nitroso. A exposição à radiação ionizante é usada para matar micróbios para esterilizar dispositivos médicos e alimentos, devido ao seu efeito dramático não específico em danificar DNA, proteínas e outros componentes celulares (veja Usando métodos físicos para controlar microorganismos).,a radiação não ionizante, como a luz ultravioleta, não é Energética o suficiente para iniciar estes tipos de alterações químicas. No entanto, a radiação não ionizante pode induzir a formação de dímero entre duas bases de pirimidina adjacentes, geralmente duas timinas, dentro de uma cadeia nucleotídica. Durante a formação de timina dímero, as duas minas adjacentes tornam-se covalentemente ligadas e, se não forem reparadas, tanto a replicação de ADN como a transcrição são bloqueadas neste ponto. A ADN polimerase pode proceder e replicar o dímero incorrectamente, levando potencialmente a mutações de mudança de fase ou de ponto.,

Figura 5. a) a radiação ionizante pode conduzir à formação de fracturas de cadeia simples e de cadeia dupla na coluna vertebral do ADN do fosfato açucareiro, bem como à modificação das bases (não apresentada). (b) a radiação não ionizante, como a luz ultravioleta, pode levar à formação de dimeros de timina, que podem impedir a replicação e transcrição e introduzir mutações de mudança de fase ou de ponto.como é que um análogo de base introduz uma mutação?,como é que um agente intercalador introduz uma mutação?que tipo de mutagénico causa dimeros de timina?
reparação do ADN
o processo de replicação do ADN é altamente exacto, mas os erros podem ocorrer espontaneamente ou ser induzidos por mutagénios. Erros não corrigidos podem levar a consequências graves para o fenótipo. As células desenvolveram vários mecanismos de reparação para minimizar o número de mutações que persistem.,A maior parte dos erros introduzidos durante a replicação do ADN são prontamente corrigidos pela maioria das polimerases do ADN através de uma função chamada “proofreading”. Na revisão, a DNA polimerase lê a base recém-adicionada, garantindo que ela é complementar à base correspondente na cadeia de template antes de adicionar a próxima. Se uma base incorreta foi adicionada, a enzima faz um corte para liberar o nucleótido errado e uma nova base é adicionada.,alguns erros introduzidos durante a replicação são corrigidos pouco depois de a máquina de replicação se ter movido. Este mecanismo é chamado reparação de desajustamento. As enzimas envolvidas neste mecanismo reconhecem o nucleótido incorretamente adicionado, excisam-no, e o substituem pela base correta. Um exemplo é o reparo de inadequação direcionado para o metil em E. coli. O ADN está hemimetilado. Isto significa que a cadeia parental é metilada enquanto a recém sintetizada cadeia filha não é. Demora vários minutos até que a nova cadeia seja metilada., Proteínas Mutl, MutL e MutH ligam-se ao local hemimetilado onde o nucleótido incorreto é encontrado. O MutH corta a cadeia não metálica (a nova cadeia). Uma exonuclease remove uma porção da cadeia (incluindo o nucleótido incorreto). A abertura formada é então preenchida por DNA pol III e ligase.
reparação de Dimeros de timina
porque a produção de dimeros de timina é comum (muitos organismos não podem evitar a luz ultravioleta), os mecanismos evoluíram para reparar estas lesões., No reparo da excisão do nucleótido (também chamado de reparação escura), as enzimas removem o dímero pirimidina e substituem-No pelos nucleótidos corretos (Figura 6). Em E. coli, o ADN é digitalizado por um complexo enzimático. Se uma distorção na dupla hélice for encontrada que foi introduzida pelo dímero pirimidina, o complexo enzimático corta a espinha dorsal de açúcar-fosfato várias bases a montante e a jusante do dímero, e o segmento de DNA entre estes dois cortes é então removido enzimaticamente. O ADN pol I substitui os nucleótidos desaparecidos pelos correctos e a ligase do ADN retém a lacuna na espinha dorsal do fosfato de açúcar.,
a reparação directa (também chamada reparação de luz) de dimeros de timina ocorre através do processo de fotorreactivação na presença de luz visível. Uma enzima chamada fotoliase reconhece a distorção na hélice de ADN causada pelo dímero de timina e liga-se ao dímero. Em seguida, na presença de luz visível, a enzima fotoliase muda de conformação e quebra o dimer de timina, permitindo que as timinas novamente a base corretamente par com as adeninas na cadeia complementar., A fotorreactivação parece estar presente em todos os organismos, à excepção dos mamíferos placentários, incluindo os seres humanos. A fotorreactivação é particularmente importante para organismos cronicamente expostos à radiação ultravioleta, como plantas, bactérias fotossintéticas, algas e corais, para prevenir a acumulação de mutações causadas pela formação de dimeros de timina.

Figura 6. Clique para obter uma imagem maior. As bactérias têm dois mecanismos para reparar dimeros de timina., a) na reparação da excisão dos nucleótidos, um complexo enzimático reconhece a distorção no complexo de ADN em torno do dímero de timina e corta e remove a cadeia de ADN danificada. Os nucleótidos corretos são substituídos por DNA pol I e a cadeia nucleótida é selada por DNA ligase. b) na fotoreactivação, a enzima fotoliase liga-se ao dímero de timina e, na presença de luz visível, separa o dímero, restabelecendo a ligação de base das timinas com adeninas complementares na cadeia de ADN oposta.,
pense nisso
- Durante a reparação do desfasamento, como é que a enzima reconhece qual é a nova e qual é a antiga Cadeia?como é que um agente intercalador introduz uma mutação?que tipo de mutação a fotoliase repara?
identificar os mutantes bacterianos
uma técnica comum utilizada para identificar os mutantes bacterianos é denominada replica plating., Esta técnica é usada para detectar mutantes nutricionais, chamados auxotróficos, que têm uma mutação num gene que codifica uma enzima na Via da biossíntese de um nutriente específico, como um aminoácido. Como resultado, enquanto as células do tipo selvagem retêm a capacidade de crescer normalmente em um meio sem o nutriente específico, os auxotróficos são incapazes de crescer em tal meio. Durante a replicação (Figura 7), uma população de células bacterianas é mutagenizada e, em seguida, banhada como células individuais em uma placa nutricionalmente completa complexa e permitida a crescer em colônias., Células destas colônias são removidas desta placa principal, muitas vezes usando veludo estéril. Este veludo, contendo células, é então pressionado na mesma orientação em placas de vários meios. Pelo menos uma placa deve ser nutricionalmente completa para garantir que as células estão sendo devidamente transferidas entre as placas. As outras placas não possuem nutrientes específicos, permitindo que o pesquisador descubra vários mutantes auxotróficos incapazes de produzir nutrientes específicos. As células da colónia correspondente na placa nutricionalmente completa podem ser utilizadas para recuperar o mutante para estudo posterior.,

Figura 7. A identificação dos mutantes auxotróficos, como os auxotróficos de histidina, é feita utilizando a placa réplica. Após mutagenese, as colónias que crescem em meio nutricionalmente completo, mas não em meio sem histidina, são identificadas como auxotróficos de histidina.porque é que as células são revestidas numa placa nutricionalmente completa, para além das placas com carência de nutrientes, quando se procura um mutante?,
O Teste de Ames
O teste de Ames, desenvolvido por Bruce Ames (1928–), na década de 1970, é um método que utiliza bactérias para uma rápida, barata análise do potencial cancerígeno de novos compostos químicos. O ensaio mede a taxa de mutação associada à exposição ao composto, que, se elevada, pode indicar que a exposição a este composto está associada a um maior risco de cancro., O teste de Ames utiliza como Organismo de ensaio uma estirpe de Salmonella typhimurium que é um auxotroph de histidina, incapaz de sintetizar a sua própria histidina devido a uma mutação num gene essencial necessário para a sua síntese. Após a exposição a um potencial mutagênico, estas bactérias são banhados em um meio carente histidina, e o número de mutantes, recuperar a capacidade de sintetizar a histidina é registrado e comparado com o número de mutantes que surgem na ausência de potencial mutagênico (Figura 8)., Os produtos químicos mais mutagénicos provocarão mais mutações com síntese de histidina restaurada no teste de Ames. Como muitos produtos químicos não são diretamente mutagênicos, mas são metabolizados em formas mutagênicas por enzimas hepáticas, extrato de fígado de rato é comumente incluído no início desta experiência para imitar o metabolismo do fígado. Após a realização do teste de Ames, os compostos identificados como mutagénicos são novamente testados quanto às suas propriedades carcinogénicas potenciais utilizando outros modelos, incluindo modelos animais como ratinhos e ratos.
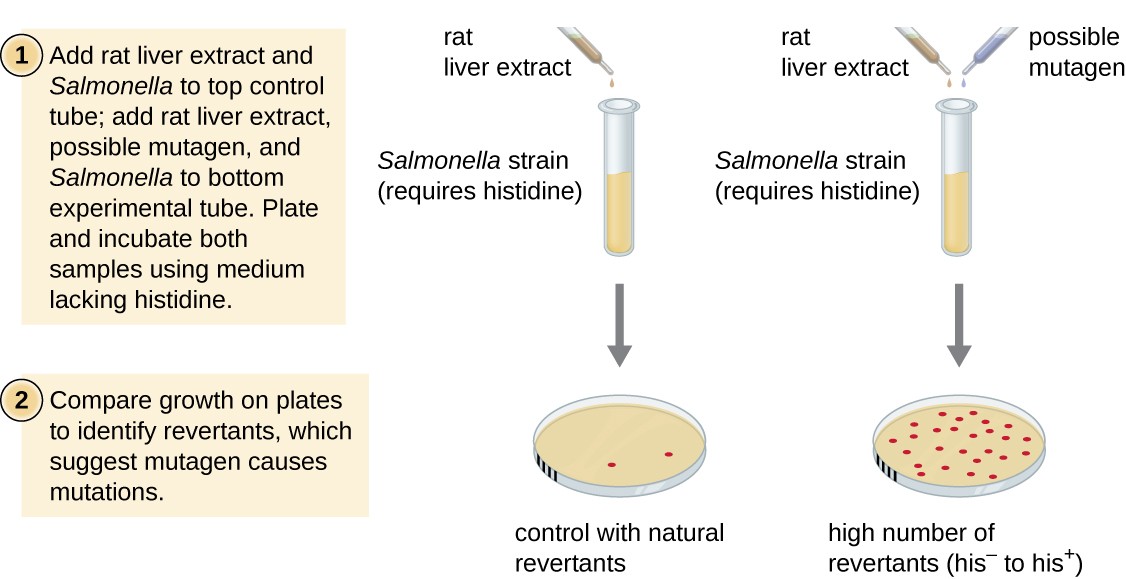
Figura 8., O teste de Ames é utilizado para identificar produtos químicos mutagénicos potencialmente carcinogénicos. A Salmonella histidina auxotroph é utilizada como estirpe de ensaio, exposta a um potencial mutagénico/carcinogénico. O número de mutantes de reversão capazes de crescer na ausência de histidina fornecido é contado e comparado com o número de mutantes de reversão natural que surgem na ausência do potencial mutagênico.que mutação é utilizada como indicador da taxa de mutação no teste de Ames?porque pode o teste de Ames funcionar como um teste de carcinogenicidade?,
conceitos-chave e resumo
- uma mutação é uma mudança hereditária no ADN. Uma mutação pode levar a uma mudança na sequência de aminoácidos de uma proteína, possivelmente afetando sua função.uma mutação pontual afecta um único par base. Um ponto de mutação pode causar uma mutação silenciosa se os códons do mRNA códigos para o mesmo aminoácido, uma mutação missense se os códons do mRNA codifica para um aminoácido diferente, ou uma mutação nonsense se os códons do mRNA torna-se um codão stop.,mutações de Missense podem manter a função, dependendo da química do novo aminoácido e da sua localização na proteína. Mutações sem sentido produzem proteínas truncadas e frequentemente não funcionais.
- uma mutação frameswift resulta de uma inserção ou eliminação de um número de nucleótidos que não é um múltiplo de três. A mudança na estrutura de leitura altera cada aminoácido após o ponto da mutação e resulta em uma proteína não funcional.mutações espontâneas ocorrem através de erros de replicação do ADN, enquanto mutações induzidas ocorrem através da exposição a um mutagénico.,os agentes mutagénicos são frequentemente carcinogénicos, mas nem sempre. No entanto, quase todos os agentes cancerígenos são mutagénicos.os mutagénicos químicos incluem análogos de base e produtos químicos que modificam bases existentes. Em ambos os casos, as mutações são introduzidas após várias rondas de replicação do ADN.radiação ionizante, como raios X e raios γ, leva à quebra da espinha dorsal do fosfodiester do DNA e também pode modificar quimicamente bases para alterar suas regras de emparelhamento base.,a radiação não ionizante, como a luz ultravioleta, pode introduzir dimeros de pirimidina (timina), que, durante a replicação e transcrição do ADN, podem introduzir mutações de frameshift ou point.as células têm mecanismos para reparar mutações que ocorrem naturalmente. A ADN polimerase tem actividade de revisão. Reparação de erros é um processo para reparar bases incorretamente incorporadas após a replicação de DNA foi concluída.os dimers da pirimidina também podem ser reparados., Em reparação de excisão nucleotídica (reparação escura), as enzimas reconhecem a distorção introduzida pelo dímero pirimidina e substituem a cadeia danificada pelas bases corretas, usando a cadeia de DNA não danificada como um modelo. Bactérias e outros organismos também podem usar reparação direta, em que a enzima fotoliase, na presença de luz visível, quebra as pirimidinas.através da comparação do crescimento na placa completa e da falta de crescimento nos meios que não possuem nutrientes específicos, podem ser identificados mutantes de perda de função específicos chamados auxotróficos.,o teste de Ames é um método pouco dispendioso que utiliza bactérias auxotróficas para medir a Mutagenicidade de um composto químico. A Mutagenicidade é um indicador do potencial carcinogénico.
escolha múltipla
Qual das seguintes é uma alteração na sequência que leva à formação de um códon de paragem?
- mutação missense
- absurdo mutação
- silencioso mutação
- exclusão de mutação
a formação de dimers pirimidina resulta de qual dos seguintes?
- espontânea erros DNA polimerase
- a exposição à radiação gama
- a exposição à radiação ultravioleta
- a exposição a intercalating agentes
Qual dos seguintes exemplos é um exemplo de mutação de frameshift?
- uma eliminação de um codão
- mutação missense
- silencioso mutação
- deleção de um nucleotídeo
Qual dos seguintes tipos de reparação do ADN em que os dimeros de timina são directamente decompostos pela enzima fotoliase?,
- directo de reparação
- nucleotide excision repair
- incompatibilidade de reparar
- revisão
Qual dos seguintes dados relativos ao teste de Ames é verdadeiro?
- It is used to identify newly formed auxotrophic mutants.é usado para identificar mutantes com atividade biossintética restaurada.é utilizado para identificar mutantes espontâneos.,
- é utilizado para identificar mutantes sem actividade de fotorreactivação.
preencha o espaço em branco
um agente mutagénico químico estruturalmente semelhante a um nucleótido, mas com regras de emparelhamento de base diferentes é chamado de ________.,
a enzima utilizada na reparação da luz para dividir os dimeros da timina é chamada_________.
o fenótipo de um organismo que é mais comumente observado na natureza é chamado de_________.,
Verdadeiro/Falso
Carcinogénios são tipicamente mutagénicos.
Pensar sobre Isso
Por que é mais provável que as inserções ou exclusões vai ser mais prejudicial para uma célula de mutações de ponto?,por que acha que o teste de Ames é preferível à utilização de modelos animais para detectar compostos químicos de mutagenicidade?
pensamento crítico
abaixo estão várias sequências de ADN que são mutadas em comparação com a sequência de tipo selvagem: 3′-T A C T G A C T G A C G A T C-5′. Visualize que cada uma é uma seção de uma molécula de DNA que se separou em preparação para a transcrição, então você só está vendo a cadeia de modelos., Construir as sequências de ADN complementares (indicando as extremidades 5′ e 3′) para cada sequência de ADN mutado, transcrever (indicando as extremidades 5′ e 3′) as cadeias template e traduzir as moléculas de ARNm utilizando o código genético, gravando a sequência de aminoácidos resultante (indicando a termini N E C). Que tipo de mutação é cada um?,>
Complementares sequência de DNA:
seqüência de mRNA transcrito do modelo:
a sequência de Aminoácidos do peptídeo:
Tipo de mutação:
Complementares sequência de DNA:
seqüência de mRNA transcrito do modelo:
a sequência de Aminoácidos do peptídeo:
Tipo de mutação: