Lernziele
- Punktmutationen und Frameshiftmutationen vergleichen
- Beschreiben Sie die Unterschiede zwischen Unsinn, Unsinn und stillen Mutationen
- Beschreiben Sie die Unterschiede zwischen Hell-und Dunkelreparatur
- Erklären Sie, wie verschiedene Mutagene wirken
- Erklären Sie, warum der Ames-Test zum Nachweis von Karzinogenen verwendet werden kann
- Analysieren Sie DNA-Sequenzen und identifizieren Sie Beispiele für Arten von Mutationen
Eine Mutation ist eine vererbbare Veränderung der DNA-Sequenz eines Organismus., Der resultierende Organismus, der als Mutant bezeichnet wird, kann im Vergleich zum Wildtyp, dem in der Natur am häufigsten beobachteten Phänotyp, eine erkennbare Veränderung des Phänotyps aufweisen. Eine Veränderung der DNA-Sequenz wird mRNA durch Transkription übertragen und kann bei Translation zu einer veränderten Aminosäuresequenz in einem Protein führen. Da Proteine die überwiegende Mehrheit der zellulären Funktionen ausführen, kann eine Änderung der Aminosäuresequenz in einem Protein zu einem veränderten Phänotyp für Zelle und Organismus führen.,
Auswirkungen von Mutationen auf die DNA-Sequenz
Es gibt verschiedene Arten von Mutationen, die nach der Veränderung des DNA-Moleküls klassifiziert werden. Ein Typ, der als Punktmutation bezeichnet wird, betrifft eine einzelne Basis und tritt am häufigsten auf, wenn eine Basis durch eine andere ersetzt oder ersetzt wird. Mutationen resultieren auch aus dem Hinzufügen einer oder mehrerer Basen, die als Insertion bezeichnet werden, oder dem Entfernen einer oder mehrerer Basen, die als Deletion bezeichnet werden.
Denken Sie darüber nach
- Welche Art von Mutation tritt auf, wenn ein Gen zwei Nukleotide weniger in seiner Sequenz hat?,
Auswirkungen von Mutationen auf Proteinstruktur und-funktion
Punktmutationen können eine Vielzahl von Auswirkungen auf die Proteinfunktion haben (Abbildung 1). Als Folge der Degeneration des genetischen Codes führt eine Punktmutation üblicherweise dazu, dass dieselbe Aminosäure trotz der Sequenzänderung in das resultierende Polypeptid eingebaut wird. Diese Veränderung hätte keinen Einfluss auf die Struktur des Proteins und wird daher als stille Mutation bezeichnet. Eine Missense-Mutation führt dazu, dass eine andere Aminosäure in das resultierende Polypeptid eingebaut wird., Die Wirkung einer Missense-Mutation hängt davon ab, wie chemisch sich die neue Aminosäure von der Wildtyp-Aminosäure unterscheidet. Die Lage der veränderten Aminosäure innerhalb des Proteins ist ebenfalls wichtig. Wenn beispielsweise die veränderte Aminosäure Teil der aktiven Stelle des Enzyms ist, kann die Wirkung der Missense-Mutation signifikant sein. Viele Missense-Mutationen führen zu Proteinen, die zumindest bis zu einem gewissen Grad noch funktionsfähig sind. Manchmal können die Auswirkungen von Missense-Mutationen nur unter bestimmten Umweltbedingungen sichtbar sein; Solche Missense-Mutationen werden als bedingte Mutationen bezeichnet., Selten kann eine Missense-Mutation von Vorteil sein. Unter den richtigen Umweltbedingungen kann diese Art von Mutation dem Organismus, der sie beherbergt, einen selektiven Vorteil verschaffen. Eine andere Art von Punktmutation, die als Unsinnmutation bezeichnet wird, wandelt ein Codon, das für eine Aminosäure kodiert (ein Sinnescodon), in ein Stop-Codon (ein Unsinn-Codon) um. Unsinnmutationen führen zur Synthese von Proteinen, die kürzer als der Wildtyp sind und typischerweise nicht funktionsfähig sind.
Löschungen und Einfügungen verursachen auch verschiedene Effekte., Da Codons Drillinge von Nukleotiden sind, können Insertionen oder Deletionen in Gruppen von drei Nukleotiden zur Insertion oder Deletion einer oder mehrerer Aminosäuren führen und keine signifikanten Auswirkungen auf die Funktionalität des resultierenden Proteins haben. Frameshift-Mutationen, die durch Einfügungen oder Löschungen einer Anzahl von Nukleotiden verursacht werden, die kein Vielfaches von drei sind, sind jedoch äußerst problematisch, da sich eine Verschiebung des Leserahmens ergibt (Abbildung 1). Da Ribosomen die mRNA in Triplecodons lesen, können Frameshift-Mutationen jede Aminosäure nach dem Mutationspunkt verändern., Der neue Leserahmen kann auch ein Stoppcodon vor dem Ende der Codierungssequenz enthalten. Folglich sind Proteine aus Genen, die Frameshift-Mutationen enthalten, fast immer nicht funktionsfähig.
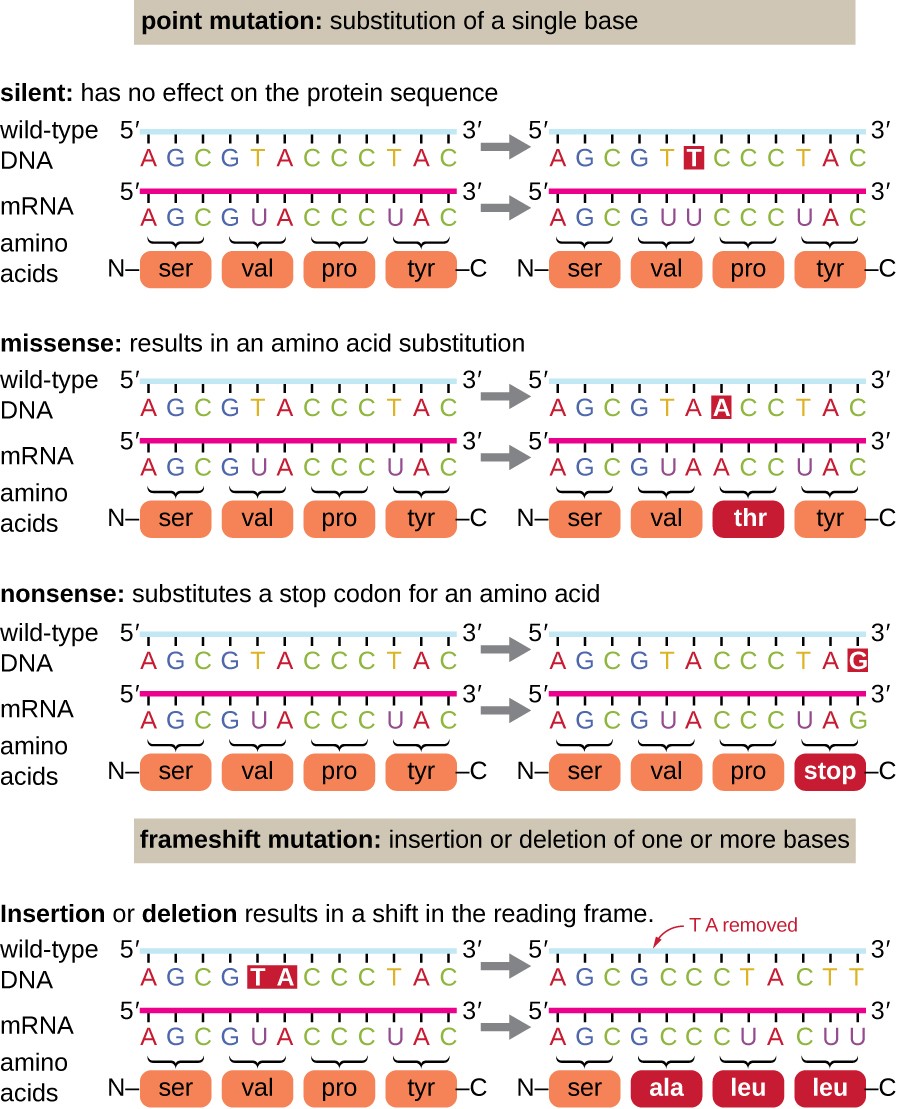
Abbildung 1. Klicken Sie für ein größeres Bild. Mutationen können zu Veränderungen in der von der DNA kodierten Proteinsequenz führen.
Denken Sie darüber nach
- Was sind die Gründe, warum eine Nukleotidänderung in einem Gen für ein Protein keinen Einfluss auf den Phänotyp dieses Gens hat?,
- Ist es möglich, dass eine Insertion von drei Nukleotiden zusammen nach dem fünften Nukleotid in ein proteinkodierendes Gen ein Protein erzeugt, das kürzer als normal ist? Wie oder wie nicht?
Eine positive Mutation
Seit 1981 der erste Fall einer Infektion mit dem humanen Immunschwächevirus (HIV) gemeldet wurde, sind fast 40 Millionen Menschen an einer HIV-Infektion gestorben, dem Virus, das das erworbene Immunschwächesyndrom (AIDS) verursacht., Das Virus zielt auf Helfer-T-Zellen ab, die eine Schlüsselrolle bei der Überbrückung der angeborenen und adaptiven Immunantwort spielen und Zellen infizieren und abtöten, die normalerweise an der Reaktion des Körpers auf Infektionen beteiligt sind. Es gibt keine Heilung für eine HIV-Infektion, aber viele Medikamente wurden entwickelt, um das Fortschreiten des Virus zu verlangsamen oder zu blockieren. Obwohl Menschen auf der ganzen Welt infiziert sein können, ist die höchste Prävalenz bei Menschen im Alter von 15 bis 49 Jahren in Afrika südlich der Sahara, wo fast jede 20.Person infiziert ist und mehr als 70% der Infektionen weltweit ausmacht (Abbildung 2)., Leider ist dies auch ein Teil der Welt, in dem Präventionsstrategien und Medikamente zur Behandlung der Infektion am meisten fehlen.

Abbildung 2. HIV ist in Afrika südlich der Sahara weit verbreitet, aber seine Prävalenz ist in einigen anderen Teilen der Welt recht gering.
In den letzten Jahren wurde wissenschaftliches Interesse durch die Entdeckung einiger Personen aus Nordeuropa geweckt, die gegen HIV-Infektionen resistent sind. Im Jahr 1998, amerikanischer Genetiker Stephen J., O ‚ Brien an den National Institutes of Health (NIH) und Kollegen veröffentlichten die Ergebnisse Ihrer genetischen Analyse von mehr als 4.000 Personen. Diese zeigten, dass viele Individuen eurasischer Abstammung (bis zu 14% in einigen ethnischen Gruppen) eine Deletionsmutation namens CCR5-delta 32 im Gen haben, das für CCR5 kodiert. CCR5 ist ein Korezeptor, der auf der Oberfläche von T-Zellen gefunden wird und für den Eintritt vieler Virusstämme in die Wirtszelle notwendig ist. Die Mutation führt zur Produktion eines Rezeptors, an den HIV nicht wirksam binden kann und blockiert somit den Viruseintritt., Menschen, die für diese Mutation homozygot sind, haben die Anfälligkeit für eine HIV-Infektion stark reduziert, und diejenigen, die heterozygot sind, haben auch einen gewissen Schutz vor Infektionen.
Es ist nicht klar, warum Menschen nordeuropäischer Abstammung diese Mutation tragen, aber ihre Prävalenz scheint in Nordeuropa am höchsten zu sein und nimmt in der Bevölkerung stetig ab, wenn man sich nach Süden bewegt. Untersuchungen zeigen, dass die Mutation seit dem Auftreten von HIV vorhanden ist und möglicherweise in europäischen Populationen aufgrund der Exposition gegenüber der Pest oder Pocken ausgewählt wurde., Diese Mutation kann Personen vor Pest (verursacht durch das Bakterium Yersinia pestis) und Pocken (verursacht durch das Variola-Virus) schützen, da dieser Rezeptor auch an diesen Krankheiten beteiligt sein kann. Das Alter dieser Mutation ist umstritten, aber Schätzungen deuten darauf hin, dass sie vor 1875 bis 225 Jahren auftrat und möglicherweise durch Wikingerinvasionen von Nordeuropa aus verbreitet wurde.
Dieser aufregende Befund hat zu neuen Wegen in der HIV-Forschung geführt, einschließlich der Suche nach Medikamenten zur Blockierung der CCR5-Bindung an HIV bei Personen, denen die Mutation fehlt., Obwohl DNA-Tests zur Bestimmung, welche Individuen die CCR5-delta 32-Mutation tragen, möglich sind, gibt es dokumentierte Fälle von Individuen, die homozygot für die Mutation sind, die sich mit HIV infiziert. Aus diesem Grund werden DNA-Tests für die Mutation von Beamten des öffentlichen Gesundheitswesens nicht allgemein empfohlen, um riskantes Verhalten bei Personen, die die Mutation tragen, nicht zu fördern. Dennoch ist die Hemmung der Bindung von HIV an CCR5 weiterhin eine gültige Strategie für die Entwicklung von medikamentösen Therapien für HIV-Infizierte.,
Ursachen von Mutationen
Fehler im Prozess der DNA-Replikation können spontane Mutationen verursachen. Die Fehlerrate der DNA-Polymerase beträgt eine falsche Base pro Milliarde replizierter Basenpaare. Die Exposition gegenüber Mutagenen kann induzierte Mutationen verursachen, bei denen es sich um verschiedene Arten chemischer Mittel oder Strahlung handelt (Tabelle 1). Die Exposition gegenüber einem Mutagen kann die Mutationsrate um das 1000-fache erhöhen. Mutagene sind oft auch Karzinogene, Mittel, die Krebs verursachen. Während jedoch fast alle Karzinogene Mutagene sind, sind nicht alle Mutagene notwendigerweise Karzinogene.,
| Tabelle 1., to AT base pair | Point | |||
|---|---|---|---|---|
| Intercalating agents | ||||
| Acridine orange, ethidium bromide, polycyclic aromatic hydrocarbons | Distorts double helix, creates unusual spacing between nucleotides | Introduces small deletions and insertions | Frameshift | |
| Ionizing radiation | ||||
| X-rays, γ-rays | Forms hydroxyl radicals | Causes single- and double-strand DNA breaks | Repair mechanisms may introduce mutations | |
| X-rays, γ-rays | Modifies bases (e.,g., Desaminieren von C nach U) | Wandelt GC in ein Basispaar um | Punkt | |
| Nichtionisierende Strahlung | ||||
| Ultraviolett | Bildet Pyrimidin (normalerweise Thymin) Dimere | Verursacht DNA-Replikationsfehler | Frameshift oder Punkt | |
Chemische Mutagene
Verschiedene Arten chemischer Mutagene interagieren direkt mit DNA, indem sie entweder als Nukleosidanaloga wirken oder Nukleotidbasen modifizieren., Chemikalien, die als Nukleosidanaloga bezeichnet werden, ähneln strukturell normalen Nukleotidbasen und können während der Replikation in die DNA integriert werden (Abbildung 3). Diese Basisanaloga induzieren Mutationen, da sie oft andere Basenpaarungsregeln haben als die Basen, die sie ersetzen. Andere chemische Mutagene können normale DNA-Basen modifizieren, was zu unterschiedlichen Basenpaarungsregeln führt. Zum Beispiel desaminiert salpetrige Säure Cytosin und wandelt es in Uracil um. Uracil paart sich dann mit Adenin in einer nachfolgenden Replikationsrunde, was zur Umwandlung eines GC-Basispaares in ein AT-Basispaar führt., Salpetrige Säure desaminiert auch Adenin zu Hypoxanthin, das Basenpaare mit Cytosin anstelle von Thymin enthält, was zur Umwandlung eines TA-Basenpaares in ein CG-Basenpaar führt.

Abbildung 3. Klicken Sie für ein größeres Bild. (a) 2-Aminopurin-Nukleosid (2AP) ist strukturell ein Nukleosid analog zu Adenin-Nukleosid, während 5-Bromouracil (5BU) ein Nukleosid analog zu Thymin-Nukleosid ist. 2AP-Basispaare mit C, Konvertieren eines AT-Basispaares in ein GC-Basispaar nach mehreren Replikationsrunden., 5. Paare mit G, Konvertieren eines AT-Basispaares in ein GC-Basispaar nach mehreren Replikationsrunden. (b) Salpetrige Säure ist eine andere Art von chemischem Mutagen, das bereits vorhandene Nukleosidbasen wie C modifiziert, um U zu erzeugen, welches Basenpaar mit A. Diese chemische Modifikation, wie hier gezeigt, führt zur Umwandlung eines CG-Basenpaares in ein TA-Basenpaar.
Chemische Mutagene, die als Interkalationsmittel bekannt sind, arbeiten unterschiedlich., Diese Moleküle gleiten zwischen den gestapelten stickstoffhaltigen Basen der DNA-Doppelhelix, verzerren das Molekül und erzeugen einen atypischen Abstand zwischen Nukleotidbasispaaren (Abbildung 4). Infolgedessen kann die DNA-Polymerase während der DNA-Replikation entweder die Replikation mehrerer Nukleotide überspringen (wodurch eine Deletion erzeugt wird) oder zusätzliche Nukleotide einfügen (wodurch eine Insertion erzeugt wird). Jedes Ergebnis kann zu einer Frameshift-Mutation führen. Verbrennungsprodukte wie polyzyklische aromatische Kohlenwasserstoffe sind besonders gefährliche Interkalationsmittel, die zu mutationsbedingten Krebserkrankungen führen können., Die Interkalationsmittel Ethidiumbromid und Acridinorange werden üblicherweise im Labor verwendet, um DNA zur Visualisierung zu färben, und sind potenzielle Mutagene.

Abbildung 4. Interkalierende Mittel, wie Acridin, führen atypische Abstände zwischen Basenpaaren ein, was dazu führt, dass DNA-Polymerase entweder eine Deletion oder eine Insertion einführt, was zu einer potenziellen Frameshift-Mutation führt.
Strahlung
Die Exposition gegenüber entweder ionisierender oder nichtionisierender Strahlung kann jeweils Mutationen in der DNA hervorrufen, wenn auch durch verschiedene Mechanismen., Starke ionisierende Strahlung wie Röntgen – und Gammastrahlen können einzel – und doppelsträngige Brüche im DNA-Rückgrat durch die Bildung von Hydroxylradikalen bei Strahlenbelastung verursachen (Abbildung 5). Ionisierende Strahlung kann auch Basen modifizieren; zum Beispiel die Desaminierung von Cytosin zu Uracil, analog zur Wirkung von Salpetersäure. Ionisierende Strahlenbelastung wird verwendet, um Mikroben abzutöten, um medizinische Geräte und Lebensmittel zu sterilisieren, aufgrund ihrer dramatischen unspezifischen Wirkung bei der Schädigung von DNA, Proteinen und anderen zellulären Komponenten (siehe Verwendung physikalischer Methoden zur Kontrolle von Mikroorganismen).,
Nichtionisierende Strahlung, wie ultraviolettes Licht, ist nicht energetisch genug, um diese Art von chemischen Veränderungen auszulösen. Nionisierende Strahlung kann jedoch eine Dimerbildung zwischen zwei benachbarten Pyrimidinbasen, üblicherweise zwei Thyminen, innerhalb eines Nukleotidstrangs induzieren. Während der Thymindimerbildung werden die beiden benachbarten Thyminen kovalent miteinander verbunden, und wenn sie nicht paariert bleiben, werden sowohl die DNA-Replikation als auch die Transkription an dieser Stelle zum Stillstand gebracht. DNA-Polymerase kann fortfahren und das Dimer falsch replizieren, was möglicherweise zu Frameshift-oder Punktmutationen führt.,

Abbildung 5. (a) Ionisierende Strahlung kann zur Bildung von einzelsträngigen und doppelsträngigen Brüchen im Zuckerphosphat-Rückgrat der DNA sowie zur Modifikation von Basen führen (nicht gezeigt). (b) Nichtionisierende Strahlung wie ultraviolettes Licht kann zur Bildung von Thymindimeren führen, die die Replikation und Transkription behindern und Frameshift-oder Punktmutationen einführen können.
Denken Sie darüber nach
- Wie führt ein Basisanalog eine Mutation ein?,
- Wie führt ein Interkalationsmittel eine Mutation ein?
- Welche Art von Mutagen verursacht Thymindimere?
DNA-Reparatur
Der Prozess der DNA-Replikation ist sehr genau, aber Fehler können spontan auftreten oder durch Mutagene induziert werden. Unkorrigierte Fehler können zu schwerwiegenden Folgen für den Phänotyp führen. Zellen haben mehrere Reparaturmechanismen entwickelt, um die Anzahl der anhaltenden Mutationen zu minimieren.,
Korrekturlesen
Die meisten Fehler, die bei der DNA-Replikation auftreten, werden von den meisten DNA-Polymerasen umgehend durch eine Funktion namens Korrekturlesen korrigiert. Beim Korrekturlesen liest die DNA-Polymerase die neu hinzugefügte Base und stellt sicher, dass sie komplementär zur entsprechenden Base im Vorlagenstrang ist, bevor sie die nächste hinzufügt. Wenn eine falsche Base hinzugefügt wurde, schneidet das Enzym, um das falsche Nukleotid freizusetzen, und es wird eine neue Base hinzugefügt.,
Mismatch Repair
Einige Fehler, die während der Replikation auftreten, werden kurz nach dem Verschieben der Replikationsmaschine behoben. Dieser Mechanismus wird als mismatch-Reparatur. Die an diesem Mechanismus beteiligten Enzyme erkennen das falsch hinzugefügte Nukleotid, verbrauchen es und ersetzen es durch die richtige Base. Ein Beispiel ist die methyl-directed mismatch repair in E. coli. Die DNA ist hemimethyliert. Dies bedeutet, dass der elterliche Strang methyliert wird, während der neu synthetisierte Tochterstrang nicht ist. Es dauert einige Minuten, bis der neue Strang methyliert ist., Proteine MutS, MutL und MutH binden an die hemimethylierte Stelle, an der das falsche Nukleotid gefunden wird. MutH schneidet den nichtmethylierten Strang (den neuen Strang). Eine Exonuklease entfernt einen Teil des Strangs (einschließlich des falschen Nukleotids). Die gebildete Lücke wird dann durch DNA pol III und Ligase gefüllt.
Reparatur von Thymindimeren
Da die Produktion von Thymindimeren häufig ist (viele Organismen können ultraviolettes Licht nicht vermeiden), haben sich Mechanismen entwickelt, um diese Läsionen zu reparieren., Bei der Nukleotid-Exzisionsreparatur (auch dunkle Reparatur genannt) entfernen Enzyme das Pyrimidin-Dimer und ersetzen es durch die richtigen Nukleotide (Abbildung 6). In E. coli wird die DNA von einem Enzymkomplex gescannt. Wenn eine Verzerrung in der Doppelhelix gefunden wird, die durch das Pyrimidindimer eingeführt wurde, schneidet der Enzymkomplex das Zuckerphosphat-Rückgrat mehrere Basen stromaufwärts und stromabwärts des Dimers, und das DNA-Segment zwischen diesen beiden Schnitten wird dann enzymatisch entfernt. DNA pol I ersetzt die fehlenden Nukleotide durch die richtigen und DNA Ligase versiegelt die Lücke im Zucker-Phosphat-Rückgrat.,
Die direkte Reparatur (auch Lichtreparatur genannt) von Thymindimeren erfolgt durch den Prozess der Photoreaktivierung in Gegenwart von sichtbarem Licht. Ein Enzym namens Photolyase erkennt die durch das Thymindimer verursachte Verzerrung in der DNA-Helix und bindet an das Dimer. Dann ändert das Photolyase-Enzym in Gegenwart von sichtbarem Licht die Konformation und zerlegt das Thymindimer, so dass die Thyminen wieder korrekt mit den Adeninen auf dem Komplementärstrang basenpaar können., Die Photoreaktivierung scheint in allen Organismen vorhanden zu sein, mit Ausnahme von Plazentasäugetieren, einschließlich Menschen. Die Photoreaktivierung ist besonders wichtig für Organismen, die chronisch ultravioletter Strahlung ausgesetzt sind, wie Pflanzen, photosynthetische Bakterien, Algen und Korallen, um die Ansammlung von Mutationen durch Thymindimerbildung zu verhindern.

Abbildung 6. Klicken Sie für ein größeres Bild. Bakterien haben zwei Mechanismen zur Reparatur von Thymindimeren., (a) Bei der Nukleotid-Exzisionsreparatur erkennt ein Enzymkomplex die Verzerrung im DNA-Komplex um das Thymindimer und schneidet und entfernt den beschädigten DNA-Strang. Die richtigen Nukleotide werden durch DNA pol I ersetzt und der Nukleotidstrang wird durch DNA Ligase versiegelt. (b) Bei der Photoreaktivierung bindet das Enzym Photolyase an das Thymindimer und bricht in Gegenwart von sichtbarem Licht das Dimer auf, wodurch die Basenpaarung der Thyminen mit komplementären Adeninen auf dem gegenüberliegenden DNA-Strang wiederhergestellt wird.,
Denken Sie darüber nach
- Wie erkennt das Enzym bei der Reparatur von Fehlanpassungen, welcher der neue und welcher der alte Strang ist?
- Wie führt ein Interkalationsmittel eine Mutation ein?
- Welche Art von Mutation repariert Photolyase?
Identifizierung bakterieller Mutanten
Eine gängige Technik zur Identifizierung bakterieller Mutanten wird als Replik-Beschichtung bezeichnet., Diese Technik wird verwendet, um Ernährungsmutanten, sogenannte Auxotrophe, zu erkennen, die eine Mutation in einem Gen aufweisen, das für ein Enzym im Biosyntheseweg eines bestimmten Nährstoffs wie einer Aminosäure kodiert. Infolgedessen, während Wildtyp-Zellen die Fähigkeit behalten, normal auf einem Medium zu wachsen, dem der spezifische Nährstoff fehlt, Auxotrophe sind nicht in der Lage, auf einem solchen Medium zu wachsen. Während des Plattierens (Abbildung 7) wird eine Population von Bakterienzellen mutagenisiert und dann als einzelne Zellen auf einer komplexen ernährungsphysiologisch vollständigen Platte plattiert und in Kolonien wachsen gelassen., Zellen aus diesen Kolonien werden von dieser Hauptplatte entfernt, oft mit sterilem Samt. Dieser Samt, der Zellen enthält, wird dann in der gleichen Ausrichtung auf Platten verschiedener Medien gepresst. Mindestens eine Platte sollte auch ernährungsphysiologisch vollständig sein, um sicherzustellen, dass die Zellen ordnungsgemäß zwischen den Platten übertragen werden. Den anderen Platten fehlen spezifische Nährstoffe, so dass der Forscher verschiedene auxotrophe Mutanten entdecken kann, die keine spezifischen Nährstoffe produzieren können. Zellen aus der entsprechenden Kolonie auf der ernährungsphysiologisch vollständigen Platte können verwendet werden, um den Mutanten zur weiteren Untersuchung wiederherzustellen.,

Abbildung 7. Die Identifizierung von auxotrophen Mutanten wie Histidin-Auxotrophen erfolgt unter Verwendung von Replikaten. Nach der Mutagenese werden Kolonien, die auf ernährungsphysiologisch vollständigem Medium, aber nicht auf Medium ohne Histidin wachsen, als Histidin-Auxotrophe identifiziert.
Denken Sie darüber nach
- Warum werden Zellen auf einer ernährungsphysiologisch vollständigen Platte zusätzlich zu nährstoffarmen Platten plattiert, wenn Sie nach einem Mutanten suchen?,
Der Ames-Test
Der von Bruce Ames (1928–) in den 1970er Jahren entwickelte Ames-Test ist eine Methode, die Bakterien zum schnellen und kostengünstigen Screening des krebserzeugenden Potenzials neuer chemischer Verbindungen verwendet. Der Test misst die Mutationsrate, die mit der Exposition gegenüber der Verbindung verbunden ist, was, falls erhöht, darauf hindeuten kann, dass die Exposition gegenüber dieser Verbindung mit einem höheren Krebsrisiko verbunden ist., Der Ames-Test verwendet als Testorganismus einen Stamm von Salmonella typhimurium, der ein Histidin-Auxotroph ist und aufgrund einer Mutation in einem für seine Synthese erforderlichen essentiellen Gen sein eigenes Histidin nicht synthetisieren kann. Nach Exposition gegenüber einem potentiellen Mutagen werden diese Bakterien auf ein Medium ohne Histidin plattiert, und die Anzahl der Mutanten, die die Fähigkeit zur Synthese von Histidin wiedererlangen, wird aufgezeichnet und mit der Anzahl solcher Mutanten verglichen, die in Abwesenheit des potentiellen Mutagens auftreten (Abbildung 8)., Chemikalien, die mutagener sind, führen im Ames-Test zu mehr Mutanten mit wiederhergestellter Histidinsynthese. Da viele Chemikalien nicht direkt mutagen sind, sondern durch Leberenzyme zu mutagenen Formen metabolisiert werden, wird Rattenleberextrakt häufig zu Beginn dieses Experiments enthalten, um den Leberstoffwechsel nachzuahmen. Nach der Durchführung des Ames-Tests werden Verbindungen, die als mutagen identifiziert wurden, mithilfe anderer Modelle, einschließlich Tiermodellen wie Mäusen und Ratten, auf ihre potenziellen krebserregenden Eigenschaften getestet.
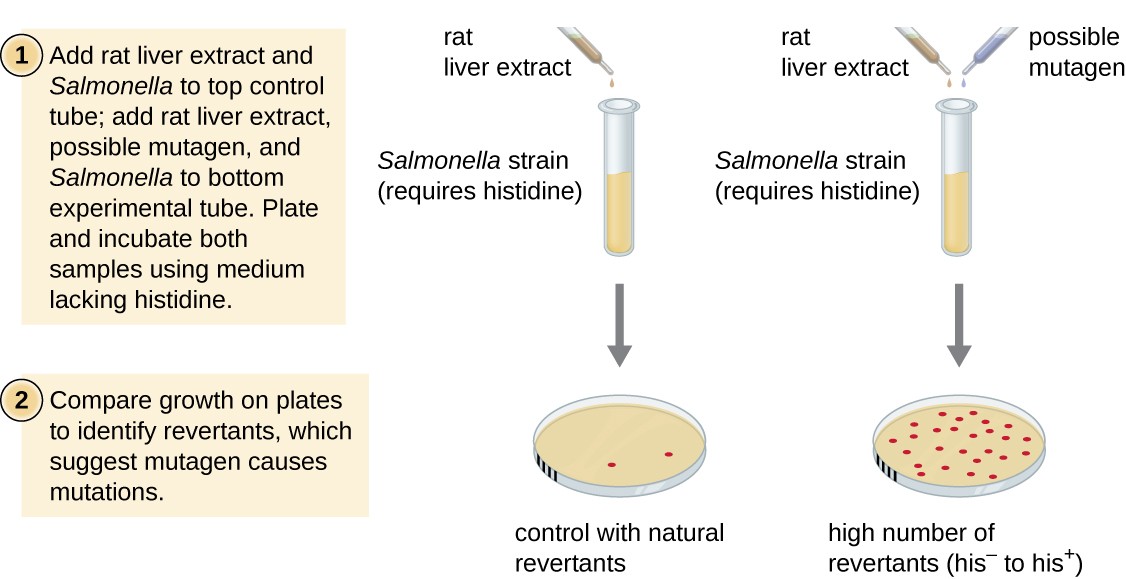
Abbildung 8., Der Ames-Test wird verwendet, um mutagene, potenziell krebserregende Chemikalien zu identifizieren. Als Teststamm wird ein Salmonella-Histidin-Auxotroph verwendet, das einem potenziellen Mutagen/Karzinogen ausgesetzt ist. Die Anzahl der Reversion-Mutanten, die in Abwesenheit von zugeführtem Histidin wachsen können, wird gezählt und mit der Anzahl der natürlichen Reversion-Mutanten verglichen, die in Abwesenheit des potentiellen Mutagens auftreten.
Denken Sie darüber nach
- Welche Mutation wird als Indikator für die Mutationsrate im Ames-Test verwendet?
- Warum kann der Ames-test als test für die Karzinogenität?,
Schlüsselkonzepte und Zusammenfassung
- Eine Mutation ist eine vererbbare Veränderung der DNA. Eine Mutation kann zu einer Veränderung der Aminosäuresequenz eines Proteins führen, die möglicherweise seine Funktion beeinflusst.
- Eine Punktmutation betrifft ein einzelnes Basispaar. Eine Punktmutation kann eine stille Mutation verursachen, wenn das mRNA-Codon für dieselbe Aminosäure kodiert, eine Unsinnmutation, wenn das mRNA-Codon für eine andere Aminosäure kodiert, oder eine Unsinnmutation, wenn das mRNA-Codon zu einem Stop-Codon wird.,
- Missense-Mutationen können ihre Funktion behalten, abhängig von der Chemie der neuen Aminosäure und ihrer Lage im Protein. Nonsense-Mutationen produzieren abgeschnittene und häufig nicht funktionierende Proteine.
- Eine Frameshift-Mutation resultiert aus einer Insertion oder Deletion einer Anzahl von Nukleotiden, die kein Vielfaches von drei ist. Die Änderung des Leserahmens verändert jede Aminosäure nach dem Mutationspunkt und führt zu einem nicht funktionierenden Protein.
- Spontane Mutationen treten durch DNA-Replikationsfehler auf, während induzierte Mutationen durch Exposition gegenüber einem Mutagen auftreten.,
- Mutagene Mittel sind häufig krebserregend, aber nicht immer. Fast alle Karzinogene sind jedoch mutagen.
- Chemische Mutagene umfassen Basenanaloga und Chemikalien, die vorhandene Basen modifizieren. In beiden Fällen werden Mutationen nach mehreren Runden der DNA-Replikation eingeführt.
- Ionisierende Strahlung wie Röntgenstrahlen und γ-Strahlen führt zum Bruch des Phosphodiester-Rückgrats der DNA und kann Basen auch chemisch modifizieren, um ihre Basenpaarungsregeln zu ändern.,
- Nichtionisierende Strahlung wie ultraviolettes Licht kann Pyrimidin – (Thymin -) Dimere einführen, die während der DNA-Replikation und-transkription Frameshift-oder Punktmutationen hervorrufen können.
- Zellen haben Mechanismen, um natürlich vorkommende Mutationen zu reparieren. DNA-Polymerase hat Korrekturleseaktivität. Mismatch Repair ist ein Prozess zur Reparatur falsch eingearbeiteter Basen, nachdem die DNA-Replikation abgeschlossen wurde.
- Pyrimidindimere können ebenfalls repariert werden., Bei der Nukleotid-Exzisionsreparatur (Dark Repair) erkennen Enzyme die durch das Pyrimidin-Dimer eingeführte Verzerrung und ersetzen den beschädigten Strang durch die richtigen Basen, wobei der unbeschädigte DNA-Strang als Schablone verwendet wird. Bakterien und andere Organismen können auch direkte Reparatur verwenden, bei der das Photolyase-Enzym in Gegenwart von sichtbarem Licht die Pyrimidine auseinanderbricht.
- Durch Vergleich des Wachstums auf der gesamten Platte und des Mangels an Wachstum auf Medien, denen bestimmte Nährstoffe fehlen, können spezifische Funktionsverlustmutanten, sogenannte Auxotrophe, identifiziert werden.,
- Der Ames-Test ist eine kostengünstige Methode, mit der auxotrophe Bakterien die Mutagenität einer chemischen Verbindung messen. Mutagenität ist ein Indikator für das karzinogene Potenzial.
Multiple Choice
Welches der folgenden ist eine Änderung der Sequenz, die zur Bildung eines Stop-Codon führt?
- nonsense mutation
- nonsense mutation
- silent mutation
- deletion mutation
Die Bildung von Pyrimidin-Dimeren ergibt sich aus welchem der folgenden?
- spontane Fehler durch DNA-Polymerase
- Exposition gegenüber Gammastrahlung
- Exposition gegenüber ultravioletter Strahlung
- Exposition gegenüber Interkalationsmitteln
Welches der folgenden Beispiele ist ein Beispiel für eine Frameshift-Mutation?
- eine Deletion eines Codons
- missense mutation
- stille Mutation
- Deletion eines Nukleotids
Welche der folgenden ist die Art der DNA-Reparatur, bei der Thymindimere direkt durch das Enzym Photolyase abgebaut werden?,
- direkte Reparatur
- Nukleotid-Exzisionsreparatur
- mismatch repair
- Korrekturlesen
Welcher der folgenden Punkte bezüglich des Ames-Tests ist wahr?
- Es wird verwendet, um neu gebildete auxotrophe Mutanten zu identifizieren.
- Es wird verwendet, um Mutanten mit wiederhergestellter biosynthetischer Aktivität zu identifizieren.
- Es wird verwendet, um spontane Mutanten zu identifizieren.,
- Es wird verwendet, um Mutanten ohne Photoreaktivierungsaktivität zu identifizieren.
Füllen Sie das leere Feld aus
Ein chemisches Mutagen, das einem Nukleotid strukturell ähnlich ist, aber unterschiedliche Basenpaarungsregeln hat, wird als ________bezeichnet.,
Das Enzym, das bei der Lichtreparatur zur Spaltung von Thymindimeren verwendet wird, heißt ________.
Der Phänotyp eines Organismus, der am häufigsten in der Natur beobachtet wird, wird ________genannt.,
True/False
Karzinogene sind in der Regel mutagen.
Denken Sie darüber nach
Warum ist es wahrscheinlicher, dass Einfügungen oder Löschungen für eine Zelle schädlicher sind als Punktmutationen?,
Warum ist Ihrer Meinung nach der Ames-Test der Verwendung von Tiermodellen vorzuziehen, um chemische Verbindungen auf Mutagenität zu untersuchen?
Kritisches Denken
Nachfolgend sind mehrere DNA-Sequenzen aufgeführt, die im Vergleich zur Wildtyp-Sequenz mutiert sind: 3′-T A C T G A C T G A C G A T C-5′. Stellen Sie sich vor, dass jeder ein Abschnitt eines DNA-Moleküls ist, das sich zur Vorbereitung der Transkription getrennt hat, sodass Sie nur den Vorlagenstrang sehen., Konstruieren Sie die komplementären DNA-Sequenzen (die 5′ und 3′ Enden anzeigen) für jede mutierte DNA-Sequenz, transkribieren Sie dann (was 5′ und 3′ Enden anzeigt) die Template-Stränge und übersetzen Sie die mRNA-Moleküle unter Verwendung des genetischen Codes, wobei Sie die resultierende Aminosäuresequenz aufzeichnen (die die N-und C-Termini angibt). Welche Art von mutation ist jeder?,>
Komplementäre DNA-Sequenz:
mRNA-Sequenz transkribiert von template:
Aminosäuresequenz des Peptids:
Typ des mutation:
Komplementäre DNA-Sequenz:
mRNA-Sequenz transkribiert aus Vorlage:
Aminosäuresequenz des Peptids:
Art der Mutation: