Struttura del cromosoma eucariotico
La lunghezza del DNA nel nucleo è molto maggiore della dimensione del compartimento in cui è contenuto. Per adattarsi a questo compartimento il DNA deve esserecondensato in qualche modo. Il grado in cui il DNA è condensato è espressocome il suo rapporto di imballaggio.
Rapporto di imballaggio-la lunghezza del DNA diviso per la lunghezza in cuiè confezionato
Ad esempio, il cromosoma umano più corto contiene 4,6 x 107 bp di DNA(circa 10 volte la dimensione del genoma di E. coli)., Ciò equivale a 14.000 µm di DNA esteso. Nel suo stato più condensato durante la mitosi, ilcromosoma è lungo circa 2 µm. Questo dà un rapporto di imballaggio di 7000 (14.000/2).
Per ottenere il rapporto di imballaggio complessivo, il DNA non viene confezionato direttamente nella struttura finale della cromatina. Invece, contiene diverse gerarchie di organizzazione.Il primo livello di imballaggio è raggiunto dall’avvolgimento del DNA attorno a un proteincore per produrre una struttura “simile a perline” chiamata nucleosoma. Thisgives un rapporto di imballaggio di circa 6., Questa struttura è invariante sia neleuchromatina che nell’eterocromatina di tutti i cromosomi. Il secondo livello di imballaggio è l’avvolgimento di perline in una struttura elicoidale chiamata fibra 30 nm che si trova sia nella cromatina interfase che nei cromosomi mitotici. Thisstructure aumenta il rapporto di imballaggio a circa 40. L’imballaggio finale si verifica quando la fibra è organizzata in loop, scaffold e domini che danno un rapporto di imballaggio finale di circa 1000 in cromosomi interfase e circa 10.000 cromosomi inmitotici.,
I cromosomi eucarioti consistono di un complesso della DNA-proteina che è organizedin un modo compatto che permette che la grande quantità di DNA sia immagazzinata nel nucleo della cellula. La designazione della subunità del cromosoma è la cromatina.L’unità fondamentale della cromatina è il nucleosoma.,romatin – l’unità di analisi del cromosoma; cromatina reflectsthe generale struttura del cromosoma, ma non è unico per ogni particularchromosome
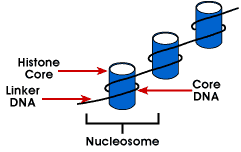
Nucleosoma – imballo più semplice struttura del DNA, che si trova inall cromosomi eucariotici; il DNA è avvolto intorno a un octamer di piccole basicproteins chiamate istoni; 146 bp si è avvolto attorno al nucleo e il remainingbases link per la prossima nucleosoma; questa struttura cause superavvolgimento negativo
Il nucleosoma è costituita da circa 200 bp, avvolto intorno a un istone octamerthat contiene due copie di istoni H2A, H2B, H3 e H4., Questi areknown come gli istoni del centro. Gli istoni sono proteine di base che hanno un’affinitàper il DNA e sono le proteine più abbondanti associate al DNA. La sequenza aminoacidica di questi quattro istoni è conservata suggerendo una funzione simile per tutti.
La lunghezza del DNA associata all’unità nucleosoma varia traspecie. Ma indipendentemente dalle dimensioni, sono coinvolti due componenti del DNA. CoreDNA è il DNA che è effettivamente associato all’ottamero dell’istone.Questo valore è invariante ed è 146 coppie di basi., Il DNA del nucleo forma due loopsaround l’ottamero, e questo permette due regioni che sono 80 bp a parte tobe portato in prossimità. Quindi, due sequenze che sono distanti possonointeragire con la stessa proteina regolatrice per controllare l’espressione genica. ILDNA che si trova tra ogni ottamero di istone è chiamato linker DNAE può variare in lunghezza da 8 a 114 coppie di basi. Questa variazione è speciesspecific, ma la variazione in lunghezza del DNA del linker egualmente è stata associata withthe lo stadio evolutivo dell’organismo o delle regioni specifiche del genoma.,
Il livello successivo di organizzazione della cromatina è la fibra a 30 nm. Questo sembra essere una struttura a solenoide con circa 6 nucleosomi per turno. Questo dà un rapporto di imballaggio di 40, il che significa che ogni 1 µm lungo l’asse contiene 40 µm di DNA. La stabilità di questa struttura richiede ilpresenza dell’ultimo membro della famiglia del gene dell’istone, l’istone H1., Perchéesperimenti che spogliano H1 dalla cromatina mantengono il nucleosoma, ma non la struttura a 30 nm, si è concluso che H1 è importante per la stabilizzazionedella struttura a 30 nm.
Il livello finale di confezionamento è caratterizzato dalla struttura di 700 nm che si vede nel cromosoma metafase. Il pezzo condensato di cromatina ha una struttura caratteristica dell’armatura che può essere rilevata nei metafasecromosomi. Ciò sembra essere il risultato di esteso ciclo del DNAin il cromosoma.
Le ultime definizioni che devono essere presentate sono euchromatina eterocromatina., Quando i cromosomi sono macchiati di coloranti, sembrano avereinternando regioni leggermente e scure. Le regioni leggermente macchiatesono euchromatin e contengono DNA geneticamente attivo a copia singola. Le regioni di Thedarkly-macchiato sono heterochromatin e contengono sequencesthat ripetitivo sono geneticamente inattive.
Centromeri e Telomeri
Centromeri e telomeri sono due caratteristiche essenziali di tutti i cromosomi eucariotici. Ciascuno fornisce una funzione unica che è assolutamente necessaria per la stabilità del cromosoma., I centromeri sono necessari per la segregazione del centromero durante la meiosi e la mitosi, ei teleomeri forniscono stabilità terminale al cromosoma e ne assicurano la sopravvivenza.
I centromeri sono quelle regioni condensate all’interno del cromosoma che sono responsabili della segregazione accurata del cromosoma replicatodurante la mitosi e la meiosi. Quando i cromosomi sono macchiati mostrano tipicamente una regione scura-macchiata che è il centromero. Durante la mitosi, il centromero che è condiviso dai cromatidi fratelli deve dividersi in modo che i cromatidi possano migrare ai poli opposti della cellula., D’altra parte, durante la prima divisione meioticail centromero dei cromatidi fratelli deve rimanere intatto, mentre durante meiosisII devono agire come fanno durante la mitosi. Pertanto il centromero è un componente importante della struttura cromosomica e della segregazione.
All’interno della regione del centromero, la maggior parte delle specie ha diverse posizioni in cui le fibre del fuso si attaccano e questi siti sono costituiti da DNA e proteine. La posizione effettiva in cui si verifica l’attaccamento è chiamata kinetochore ed è composta sia da DNA che da proteine. Viene chiamata la sequenza del DNA all’interno di queste regionidagnato cinese., Poiché il DNA CEN può essere spostato da un cromosoma all’altro e fornire ancora al cromosoma la capacità di segregare, queste sequenze non devono fornire alcuna altra funzione.
Tipicamente il DNA CEN è lungo circa 120 coppie di basi e consiste di diversi sottodomini, CDE-I, CDE-II e CDE-III. Le mutazioni nei primi due sottodomini non hanno alcun effetto sulla segregazione, ma una mutazione puntuale nel sottodominio CDE-III elimina completamente la capacità del centromero di funzionare durante la segregazione cromosomica., Pertanto CDE-III deve essere attivamente coinvolto nel legame delle fibre del mandrino al centromero.
La componente proteica del kinetochore viene solo ora caratterizzata. Un complesso di tre proteine chiamato Cbf-III si lega alle normali regioni CDE-III ma non può legarsi a una regione CDE-III con una mutazione puntiforme che impedisce la segregazione mitotica. Inoltre, i mutanti dei geni che codificano le proteine Cbf-III eliminano anche la capacità dei cromosomi di segregare durante la mitosi., Ulteriori analisi del DNA e dei componenti proteici del centromero sono necessarie per comprendere appieno la meccanica della segregazione cromosomica.
I telomeri sono la regione del DNA alla fine del cromosoma eucariotico lineare che sono necessari per la replicazione e la stabilità del cromosoma.McClintock riconobbe le loro caratteristiche speciali quando notò, che se due cromosomi fossero rotti in una cella, la fine di uno potrebbe attaccarsi all’altroe viceversa. Ciò che non ha mai osservato è stato l’attaccamento del brokenend alla fine di un cromosoma ininterrotto., Così le estremità dei cromosomi spezzati sono appiccicose, mentre l’estremità normale non è appiccicosa, suggerendo che le estremità ofchromosomes hanno caratteristiche uniche. Di solito, ma non sempre, il telomericDNA è eterocromatico e contiene sequenze dirette ripetute in tandem. La seguente tabella mostra le sequenze di ripetizione di diverse specie. Questi sono spesso della forma (T/A)xGy dove x è tra 1 e 4 e y è maggiore di 1.,
Telomere Repeat Sequences
| Species | Repeat Sequence |
|---|---|
| Arabidopsis | TTTAGGG |
| Human | TTAGGG |
| Oxytricha | TTTTGGGG |
| Slime Mold | TAGGG |
| Tetrahymena | TTGGGG |
| Trypanosome | TAGGG |
| Yeast | (TG)1-3TG2-3 |
Notice that the number of TG sequences and the number of cytosines in the yeast sequence varies., Almeno per il lievito, è stato dimostrato che diversi ceppi contengono diverse lunghezze di teleomeri e che la lunghezza è sotto controllo genetico.
La difficoltà primaria con i telomeri è la replicazione del filamento in ritardo.Poiché la sintesi del DNA richiede un modello di RNA (che fornisce il libero 3 ‘ – OHgroup) per innescare la replicazione del DNA, e questo modello alla fine viene degradato, una breve regione a filamento singolo verrebbe lasciata alla fine del cromosoma. Questa regione sarebbe suscettibile agli enzimi che degradano il DNA a filamento singolo., Il risultato sarebbe che la lunghezza del cromosoma sarebbe accorciata dopo ogni divisione. Ma questo non si vede.
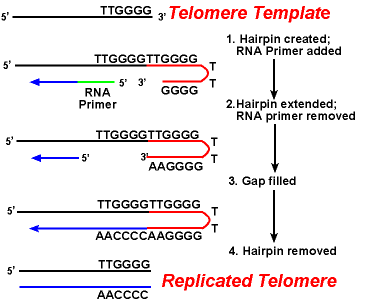
L’azione degli enzimi telomerasi assicura che le estremità dei fili in ritardo siano replicate correttamente. Un sistema ben studiato coinvolge l’organismo dei protozoi Tetrahymena. I telomeri di questo organismo terminano nella sequenza 5 ‘- TTGGGG-3’. La telomerasi aggiunge una serie di ripetizioni 5′-TTGGGG-3′ alle estremità del filo in ritardo. Una forcina si verifica quando coppie di basi insolite tra residui di guanina nella forma ripetuta., Successivamente il primer RNA viene rimosso e l’estremità 5′ del filo in ritardo può essere utilizzata per la sintesi del DNA. La legatura avviene tra il filo in ritardo finito e la forcina. Infine, il tornante viene rimosso alla ripetizione 5 ‘- TTGGGG-3’. Così la fine del cromosoma è fedelmente replicata. La figura seguente mostra questi passaggi.,
La Replicazione dei Telomeri
l’Analisi di Sequenze di DNA in Genomi Eucariotici
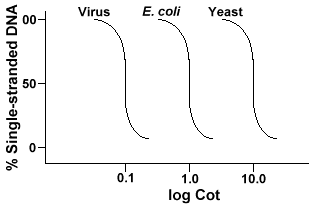
La tecnica che viene utilizzata per determinare la sequenza complessità di qualsiasi genomeinvolves la denaturazione e rinaturazione del DNA. Il DNA è denaturedby riscaldamento che scioglie i legami H e rende il DNA single-stranded. Seil DNA viene rapidamente raffreddato, il DNA rimane a filamento singolo. Ma se il DNAis permesso di raffreddare lentamente, sequenze che sono complementari troveranno vicenda e alla fine coppia di base di nuovo., La velocità con cui il DNA reanneals (un altro termine per renature) è una funzione della specie da cui il DNAwas isolato. Di seguito è riportata una curva ottenuta da un semplice genoma.
L’asse Y è la percentuale del DNA che rimane single stranded. Questo èespresso come un rapporto tra la concentrazione di DNA a filamento singolo (C)e la concentrazione totale del DNA iniziale (Co). L’asse X èuna scala log del prodotto della concentrazione iniziale di DNA (in moli/litro)moltiplicata per il tempo in cui la reazione è proceduta (in secondi)., La designazione di questo valore è Cot ed è chiamata valore “Cot”. La curva stessaè chiamata curva “Culla”. Come si può vedere la curva è piuttosto liscia che indica che la riannealing si verifica rallentando ma gradualmente in un periodo di tempo. Un valore particolare che è utile è Cot½, theCot valore in cui la metà del DNA ha reannealed.
Passi coinvolti negli esperimenti di denaturazione e rinaturazione del DNA
1. Taglia il DNA a una dimensione di circa 400 bp.
2. Denaturare il DNA riscaldando a 100oC.
3. Raffreddare lentamente e prelevare campioni a intervalli di tempo diversi.
4., Determinare la % di DNA a singolo filamento in ogni punto temporale.
La forma di una “Culla” della curva per una determinata specie, è una funzione di due fattori:
- dimensioni o complessità del genoma; e
- la quantità di DNA ripetitivo all’interno del genoma
Se tracciamo la “Culla” delle curve del genoma di tre specie come bacteriophagelambda, E. coli e lievito di birra, vedremo che essi hanno la stessa forma,ma il Cot½ del lievito sarà più grande, E. coli nextand lambda più piccolo., Fisicamente, più grande è la dimensione del genoma il itwill più lungo prende affinchè tutta una sequenza incontri la sua sequenza complementare nella soluzione. Questo perché due sequenze complementari devono incontrarsi l’un l’altro prima che possano accoppiarsi. Il più complesso il genoma, che è themore sequenze uniche che sono disponibili, più tempo ci vorrà per anytwo sequenze complementari di incontrarsi e coppia. Dato similarconcentrations in soluzione, allora prenderà una specie più complessa longper raggiungere Cot½ .,
Sequenze di DNA ripetute, sequenze di DNA che si trovano più di una volta inil genoma della specie, hanno effetti distintivi sulle curve “Cot”.Se una sequenza specifica è rappresentata due volte nel genoma, avrà due sequenze complementari con cui accoppiarsi e come tale avrà un valore Cot grande la metà di una sequenza rappresentata solo una volta nel genoma.
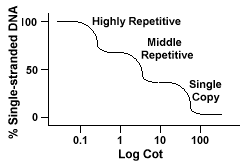
I genomi eucariotici hanno in realtà una vasta gamma di sequenze che sono rappresentatea diversi livelli di ripetizione., Le sequenze di copia singola si trovano una voltao alcune volte nel genoma. Molte delle sequenze che codificano functionalgenes rientrano in questa classe. Il DNA ripetitivo medio si trova da 10s -1000 volte nel genoma. Esempi di questi includono rRNA e TRNAGENI e proteine di stoccaggio in piante come il mais. Il DNAC ripetitivo medio può variare da 100-300 bp a 5000 bp e può essere disperso in tutto il genoma.Le sequenze più abbondanti si trovano nel DNAclass altamente ripetitivo., Queste sequenze si trovano da 100.000 a 1 milione di volte nel genomee possono variare in dimensioni da poche a diverse centinaia di basi di lunghezza. Thesesequences sono trovati nelle regioni del cromosoma quali l’eterocromatina, i centromeri ed i telomeri e tendono ad essere sistemati come ripetizioni di tandem. Thefollowing è un esempio di una sequenza ripetuta in tandem:
ATTATA ATTATA ATTATA / /ATTATA
Genomi che contengono queste diverse classi di sequenze reanneal in un differentmanner rispetto ai genomi con solo sequenze di copia singola., Invece di avere una singola curva “Cot” liscia, si possono vedere tre curve distinte, ognuna delle quali rappresenta una diversa classe di ripetizione. Le prime sequenze a reanneal sono le sequenze highlyrepetitive perché così tante copie di loro esistono nel genoma, e perché hanno una complessità di sequenza bassa. La seconda porzione di thegenome a reanneal è il DNA ripetitivo medio, e la porzione finale toreanneal è il DNA singola copia., Il seguente diagramma mostra la curva ” Cot “per un genoma eucariotico “tipico”
La seguente tabella fornisce la distribuzione della sequenza per le specie selezionate.
| Specie | Distribuzione della sequenza |
|---|---|
| Batteri | 99.,27% Medio Ripetitivo 8% Altamente Ripetitivo |
| Mais | 30% Copia Singola 40% Medio Ripetitivo 20% Altamente Ripetitivo |
| Grano | 10% Singola Copia 83% Medio Ripetitivo 4% Altamente Ripetitivo |
| Arabidopsis | 55% a Copia Singola 27% Medio Ripetitivo 10% Altamente Ripetitivo |
Sequenza Interspersion
Anche se i genomi di organismi superiori contenere copia singola, middlerepetitive e altamente ripetitiva di sequenze di DNA, queste sequenze non sono arrangedsimilarly in tutte le specie., La disposizione prominente è chiamata breve periodointerspersione. Questa disposizione è caratterizzata da sequenze ripetute100-200 bp di lunghezza intervallati tra sequenze a copia singola che sono 1000-2000bp di lunghezza. Questa disposizione si trova in animali, funghi e piante.
Il secondo tipo di disposizione è l’interspersione a lungo periodo. Questo è caratterizzato da 5000 bp tratti di sequenze ripetute intervallate all’interno di regioni di 35.000 bp di DNA a singola copia. Drosophila è un esempio di una specie con questa disposizione sequenza non comune., In entrambi i casi, le sequenze ripetute provengono solitamente dalla classe media ripetitiva. Abbiamo discusso sopra dove si trovano sequenze altamente ripetitive.
Cariotipo del cromosoma eucariotico
Mentre i batteri hanno un solo cromosoma, le specie eucariotiche hanno almeno una coppia di cromosomi. La maggior parte hanno più di una coppia. Un altro punto rilevante è che i cromosomi eucariotici vengono rilevati solo durante la divisione cellulare e non durante tutte le fasi del ciclo cellulare. Sono nella loro forma mostcondensed durante la metafase quando i cromatidi della sorella sono attaccati.,Questo è lo stadio primario in cui viene eseguita l’analisi citogenetica.
Ogni specie è caratterizzata da un cariotipo. Il cariotipo è adescrizione del numero di cromosomi nella normale cellula diploide, comecome la loro distribuzione dimensionale. Ad esempio, il cromosoma umano ha 23 coppiedi cromosoma, 22 coppie somatiche e una coppia di cromosomi sessuali. Un aspetto importante della ricerca genetica è la correlazione dei cambiamenti nel cariotipo con i cambiamenti nel fenotipo dell’individuo.
Un aspetto importante della genetica è la correlazione dei cambiamenti nel cariotipo con i cambiamenti nel fenotipo., Ad esempio, gli esseri umani che hanno un cromosoma extra 21hanno la sindrome di Down. Inserzioni, delezioni e cambiamenti nel numero cromosomicopuò essere rilevato dal citogenetista esperto, ma correlare questi fenotipi specifici è difficile.
Il primo parametro discriminante quando si sviluppa un cariotipo è la dimensionee il numero dei cromosomi. Sebbene ciò sia utile, non fornisce dettagli sufficienti per iniziare lo sviluppo di una correlazione tra struttura e funzione (fenotipo)., Per distinguere ulteriormente tra i cromosomi, lorosono trattati con un colorante che colora il DNA in modo riproducibile. Afterstaining, alcune delle regioni sono leggermente macchiate ed altre sono heavilystained. Come descritto sopra, le regioni leggermente macchiate sono calledeuchromatin e la regione macchiata scura è calledheterochromatin. Il colorante corrente di chose è la macchia Giemsa, e il modello risultante è chiamato il modello G-banding.,
Paradosso del valore C
Oltre a descrivere il genoma di un organismo per il suo numero di cromosomi,è anche descritto dalla quantità di DNA in una cellula aploide. Questo è solitamente espresso come la quantità di DNA per cellula aploide (solitamente espressa come picogrammi)o il numero di kilobasi per cellula aploide ed è chiamato il valore C. Una caratteristica immediata degli organismi eucariotici evidenzia un’anomalia specifica che è stata rilevata all’inizio della ricerca molecolare., Anche se gli organismi eucarioti sembrano avere 2-10 volte più geni dei procarioti, hanno molti ordini di grandezza più DNA nella cellula. Inoltre, la quantità di DNA per genomeis correlato non con la presunta complessità evolutiva di una specie.Questo è indicato come il paradosso del valore C: la quantità di DNA nella cellula aploidica di un organismo non è correlata alla sua complessità evolutiva. (Un altro punto importante da tenere a mente è che non esiste alcuna relazione tra il numero di cromosomi e la presunta complessità evolutiva di un organismo.,)
C Values of Organisms Used in Genetic Studies
| Species | Kilobases/haploid genome |
|---|---|
| E. coli |
4.5 x 103 |
| Human |
3.0 x 106 |
| Drosophila |
1.7 x 105 |
| Maize |
2.0 x 106 |
| Aribidopsis |
7.,0 x 104 |
Un esempio drammatico dell’intervallo di valori C può essere visto nel regno dell’impiantodove Arabidopsis rappresenta la fascia bassa e lily (1.0 x 10^8 kb/haploidgenome) la fascia alta della complessità. In termini di peso questo è 0.07 picogramsper aploide Arabidopsis genoma e 100 picogrammi per aploide lilygenome.
Genoma-l’insieme completo di cromosomi ereditati da un singolo genitore;la componente completa del DNA di un individuo; la definizione spesso escludeorganelli