eukariotyczna struktura chromosomu
Długość DNA w jądrze jest znacznie większa niż wielkość kompartmentyny, w której się znajduje. Aby zmieścić się w tym przedziale, DNA musi być w jakiś sposób licencjonowane. Stopień, w jakim DNA jest skondensowane wyraża sięjako jego stosunek pakowania.
stosunek pakowania-Długość DNA podzielona przez długośćjest pakowany
na przykład najkrótszy ludzki chromosom zawiera 4,6 x 107 bp DNA(około 10 razy rozmiar genomu E. coli)., Odpowiada to 14 000 µm rozszerzonego DNA. W stanie najbardziej skondensowanym podczas mitozy, chromosom ma około 2 µm Długości. Daje to stosunek pakowania 7000(14,000/2).
aby osiągnąć ogólny stosunek opakowania, DNA nie jest pakowane bezpośrednio do końcowej struktury chromatyny. Zamiast tego zawiera kilka hierarchii organizacji.Pierwszy poziom pakowania jest osiągany przez nawijanie DNA wokół proteincore, aby wytworzyć strukturę” przypominającą koralik ” zwaną nukleosomem. To daje stosunek pakowania około 6., Struktura ta jest niezmienna zarówno w teuchromatynie, jak i heterochromatynie wszystkich chromosomów. Drugi poziom opakowaniajest zwijanie koralików w spiralnej strukturze zwanej włóknem 30 nm, który znajduje się zarówno w chromatynie międzyfazowej, jak i chromosomach mitotycznych. Ta struktura zwiększa stosunek pakowania do około 40. Końcowe pakowanie występuje, gdy włókno jest zorganizowane w pętle, rusztowania i domeny, które dają współczynnik końcowego pakowania około 1000 w chromosomach międzyfazowych i około 10 000 chromosomów inmitotycznych.,
chromosomy eukariotyczne składają się z kompleksu DNA-białka, który jest zorganizowany w zwarty sposób, który pozwala na przechowywanie dużej ilości DNA w jądrze komórki. Podjednostką chromosomu jest chromatyna.Podstawową jednostką chromatyny jest nukleosom.,romatyna – Jednostka analizy chromosomu; chromatyna odzwierciedla ogólną strukturę chromosomu, ale nie jest unikalna dla żadnego partiarchromosomu
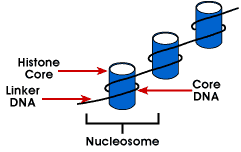
Nucleosome – najprostsza struktura opakowaniowa DNA, która znajduje się we wszystkich chromosomach eukariotycznych; DNA jest owinięte wokół oktameru małych bazowych protein zwanych histonami; 146 bp jest owinięty wokół rdzenia, a pozostałe komórki łączą się z następnym nukleosomem; struktura ta powoduje ujemne nadkrycie
nukleosom składa się z około 200 BP owinięty wokół oktameru histonowego, który zawiera dwie kopie białek histonowych H2A, H2B, H3 i H4., Są one znane jako histony rdzenia. Histony są podstawowymi białkami, które mają powinowactwo do DNA i są najbardziej obfitymi białkami związanymi z DNA. Sekwencja aminowa tych czterech histonów jest zachowana, co sugeruje podobną funkcję dla wszystkich.
Długość DNA związana z jednostką nukleosomu różni się w zależności od gatunku. Ale niezależnie od wielkości, dwa składniki DNA są zaangażowane. CoreDNA jest DNA, które jest faktycznie związane z oktamerem histonu.Wartość ta jest niezmienna i wynosi 146 par bazowych., Rdzeń DNA tworzy dwie pętle wokół oktameru, co pozwala na zbliżenie dwóch regionów oddalonych od siebie o 80 bp. Tak więc, dwa sekwencje które są daleko od siebie mogą współdziałać z tym samym białkiem regulacyjnym kontrolować ekspresję genu. DNA, który znajduje się pomiędzy każdym oktamerem histonu, nazywa się dna łącznika i może różnić się długością od 8 do 114 par zasad. Ta zmienność jest specyficzna gatunkowo, ale zmienność długości DNA łącznika jest również związana z etapem rozwojowym organizmu lub określonymi regionami genomu.,
kolejnym poziomem organizacji chromatyny jest włókno 30 nm. Jest to struktura solenoidowa z około 6 nukleosomami na turę. To daje stosunek opakowania 40, co oznacza, że co 1 µm wzdłuż osi zawiera 40 µm DNA. Stabilność tej struktury wymaga obecności ostatniego członka rodziny genów histonowych, histonu H1., Z uwagi na fakt, że ekstrakty oddzielające H1 od chromatyny utrzymują nukleosom, ale nie strukturę 30 nm, stwierdzono, że H1 jest ważny dla stabilizacji struktury 30 nm.
końcowy poziom opakowania charakteryzuje się strukturą 700 nm widzianą w chromosomie metafazy. Skondensowany fragment chromatyny ma arakterystyczną strukturę rusztowania, którą można wykryć w metafazechromosomach. Wydaje się to być wynikiem rozległego zapętlenia DNA chromosomu.
Ostatnie definicje, które należy przedstawić to euchromatyna iheterochromatyna., Gdy chromosomy są zabarwione barwnikami, wydają się mieć lekko i ciemno zabarwione obszary. Słabo zabarwione regiony są euchromatyną i zawierają pojedynczą kopię, genetycznie aktywnego DNA. Obszary te są heterochromatyną i zawierają powtarzające się sekwencje, które są genetycznie nieaktywne.
Centromery i telomery
Centromery i telomery są dwiema istotnymi cechami wszystkich chromosomów eukariotycznych. Każdy zapewnia unikalną funkcję, która jest absolutnie niezbędna dla stabilności chromosomu., Centromery są potrzebne do segregacji centromeru podczas mejozy i mitozy, a teleomery zapewniają stabilność terminalną chromosomu i zapewniają jego przetrwanie.
Centromery to te skondensowane regiony w obrębie chromosomu, które są odpowiedzialne za dokładną segregację replikowanego chromosomu w trakcie mitozy i mejozy. Gdy chromosomy są zabarwione, zazwyczaj pokazują ciemny obszar, który jest centromerem. Podczas mitozy centromer dzielony przez chromatydy siostrzane musi się dzielić, aby chromatydy mogły migrować do biegunów komórki., Z drugiej strony, podczas pierwszego podziału mejotycznegocentromery chromatyd siostrzanych muszą pozostać nienaruszone, natomiast podczas mejozy muszą działać tak, jak podczas mitozy. Dlatego centromer jest ważnym składnikiem struktury i segregacji chromosomów.
w obrębie regionu centromeru większość gatunków ma kilka miejsc, w których przyłączają się włókna wrzecionowate, a miejsca te składają się zarówno z DNA, jak i białka. Rzeczywiste miejsce, w którym następuje przyłączenie, nazywa się kinetochorem i składa się zarówno z DNA, jak i białka. Sekwencja DNA w obrębie tych regionów nazywa się DNA cen., Ponieważ DNA CEN może być przenoszone z jednego chromosomu do drugiego i nadal zapewnia chromosomowi zdolność do segregacji, sekwencje te nie mogą zapewniać żadnej innej funkcji.
zazwyczaj CEN DNA ma około 120 par zasad i składa się z kilku sub-domen, CDE-i, CDE-II i CDE-III. mutacje w dwóch pierwszych sub-domenach nie mają wpływu na segregację, ale mutacja punktowa w sub-domenie CDE-III całkowicie eliminuje zdolność centromeru do funkcjonowania podczas segregacji chromosomów., Dlatego CDE-III musi aktywnie uczestniczyć w wiązaniu włókien wrzeciona z centromerem.
dopiero teraz scharakteryzowano białkowy Składnik kinetochoru. Kompleks trzech białek zwanych CBF-III wiąże się z normalnymi regionami CDE-III, ale nie może wiązać się z regionem CDE-III z mutacją punktową, która zapobiega segregacji mitotycznej. Ponadto mutanty genów kodujących białka Cbf-III eliminują również zdolność do segregacji chromosomów podczas mitozy., Dodatkowe analizy DNA i składników białkowych centromeru są konieczne, aby w pełni zrozumieć mechanikę segregacji chromosomów.
telomery są regionem DNA na końcu liniowego eukariotychromosomu, które są wymagane do replikacji i stabilności chromosomu.McClintock rozpoznała ich szczególne cechy, gdy zauważyła, że jeśli dwaochromosomy zostały rozbite w komórce, koniec jednego z nich może przyczepić się do drugiego i odwrotnie. Nigdy nie zauważyła przywiązania złamanego do końca nieprzerwanego chromosomu., Tak więc końce złamanych chromosomów są lepkie, podczas gdy normalny koniec nie jest lepki, co sugeruje, że końce chromosomów mają unikalne cechy. Zwykle, ale nie zawsze, telomericDNA jest heterochromatyczna i zawiera bezpośrednie, tandemowo powtarzające się sekwencje. Poniższa tabela przedstawia sekwencje powtórzeń kilku gatunków. Często mają one postać (T/A)xGy, gdzie x jest między 1 A 4, A y jest większe niż 1.,
Telomere Repeat Sequences
| Species | Repeat Sequence |
|---|---|
| Arabidopsis | TTTAGGG |
| Human | TTAGGG |
| Oxytricha | TTTTGGGG |
| Slime Mold | TAGGG |
| Tetrahymena | TTGGGG |
| Trypanosome | TAGGG |
| Yeast | (TG)1-3TG2-3 |
Notice that the number of TG sequences and the number of cytosines in the yeast sequence varies., Przynajmniej w przypadku drożdży wykazano, że różne szczepy zawierają różne długości teleomerów i że długość jest pod kontrolą genetyczną.
podstawową trudnością w przypadku telomerów jest replikacja pasma opóźniającego.Ponieważ synteza DNA wymaga szablonu RNA (który zapewnia wolną grupę 3 ' – OH) do replikacji DNA, a ten szablon jest ostatecznie zdegradowany, krótki jednoniciowy region pozostałby na końcu chromosomu. Region ten byłby podatny na enzymy, które degradują jednoniciowe DNA., W rezultacie długość chromosomu uległaby skróceniu po każdym podziale. Ale tego nie widać.
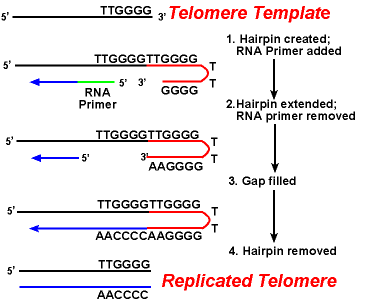
działanie enzymów telomerazy zapewnia prawidłową replikację końców opóźnionych nici. Dobrze zbadany system obejmuje organizm pierwotniaków Tetrahymena. Telomery tego organizmu kończą się sekwencją 5 '- TTGGGG-3′. Telomeraza dodaje serię 5′-TTGGGG-3 ' powtarza się na końcach pasma opóźniającego. Spinka do włosów występuje, gdy nietypowe pary zasad między reszt guaniny w postaci powtórzeń., Następnie usuwa się podkład RNA, a 5 ' koniec opóźnionej nici może być użyty do syntezy DNA. Ligacja występuje między gotowym pasem lagging i Spinka do włosów. Wreszcie, spinka jest usuwany w 5′ – ttgggg-3 ' powtórzyć. W ten sposób koniec chromosomu jest wiernie replikowany. Poniższy rysunek pokazuje te kroki.,
replikacja telomerów
analiza sekwencji DNA w genomach eukariotycznych
technika używana do określenia złożoności sekwencji dowolnego genomu obejmuje denaturację i renaturacja DNA. DNA jest denaturowane przez ogrzewanie, które topi wiązania H i sprawia, że DNA jednoniciowy. Jeśli DNA jest szybko chłodzony, DNA pozostaje jednoniciowy. Ale jeśli DNAis pozwoli schłodzić się powoli, sekwencje, które są komplementarne, znajdą się wzajemnie i ostatecznie ponownie para bazowa., Szybkość, z jaką DNA reanneals (inny termin na renature) jest funkcją gatunku, z którego DNAwas wyizolowane. Poniżej znajduje się krzywa, która jest otrzymywana z prostego genomu.
oś Y to procent DNA, który pozostaje jednoniciowy. Jest to wyrażone jako stosunek stężenia jednoniciowego DNA (C) do całkowitego stężenia wyjściowego DNA (Co). W osi X log-skala iloczynu początkowego stężenia DNA (w Mol/litr)pomnożona przez czas trwania reakcji (w sekundach)., Oznaczenie dla tej wartości jest Cot i nazywa się wartością „Cot”. Sama krzywa nazywa się krzywą „Cot”. Jak widać, krzywa jest raczej gładka, co wskazuje, że ponowne nagrzewanie następuje spowolnienie, ale stopniowo w okresie czasu. Jedną szczególną wartością, która jest użyteczna, jest Cot½ , wartość kot, w której połowa DNA została ponownie uszczelniona.
etapy denaturacji i Renaturacji DNA
1. Ściąć DNA do wielkości około 400 bp.
2 . Denature DNA przez ogrzewanie do 100oC.
3. Powoli schłodzić i pobierać próbki w różnych odstępach czasu.
4., Określ % jednoniciowego DNA w każdym punkcie czasowym.
kształt krzywej „Cot” dla danego gatunku jest funkcją dwóch czynników:
- wielkość lub złożoność genomu; i
- ilość powtarzalnego DNA w genomie
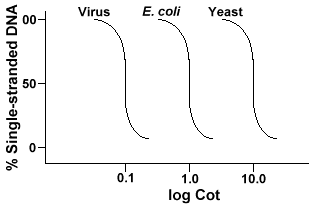
Jeśli wykreślimy krzywe „Cot” genomu trzech gatunków, takich jak bakteriofagelambda,E. coli i drożdże, zobaczymy, że mają ten sam kształt, ale Cot½ drożdży będzie największy, E. coli.coli i lambda najmniejsze., Fizycznie, im większy rozmiar genomu, tym dłużej zajmie, aby jedna sekwencja napotkała jego komplementarną sekwencję w roztworze. Dzieje się tak, ponieważ dwie komplementarne sekwencje muszą napotykaćkażdy inny, zanim będą mogły się sparować. Im bardziej złożony Genom, tzn. więcej unikalnych sekwencji, które są dostępne, tym dłużej będzie potrzeba, aby dowolne dwie komplementarne sekwencje spotkały się ze sobą i sparowały. Biorąc pod uwagę podobne koncentracje w roztworze, zajmie to bardziej złożonemu gatunkowi długo dotarcie do co½ .,
powtarzane sekwencje DNA, sekwencje DNA, które znajdują się więcej niż raz w genomie gatunku, mają charakterystyczny wpływ na krzywe „Cot”.Jeśli określona sekwencja jest reprezentowana dwa razy w genomie, będzie miała dwie sekwencje uzupełniające do parowania i jako taka będzie miała wartość Cot o połowę większą niż Sekwencja reprezentowana tylko raz w genomie.
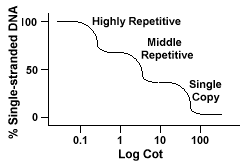
genomy eukariotyczne mają w rzeczywistości szeroki wachlarz sekwencji, które są reprezentowane na różnych poziomach powtórzeń., Pojedyncze sekwencje kopii znajdują się jednokrotnielub kilka razy w genomie. Wiele sekwencji kodujących geny funkcyjne należy do tej klasy. Środkowe powtarzalne DNA znajdują się od 10s -1000 razy w genomie. Przykładami mogą być rRNA i trnageny oraz białka magazynujące w roślinach takich jak kukurydza. Średni powtarzalny DNAcan waha się od 100-300 bp do 5000 bp i może być rozproszony w całym genomie.Najliczniejsze sekwencje występują w silnie powtarzalnym Dnieprze., Sekwencje te występują od 100 000 do 1 miliona razy w genomie i mogą wahać się od kilku do kilkuset baz długości. Te sekwencje znajdują się w regionach chromosomu, takich jak heterochromatyna, centromery i telomery i wydają się być ułożone jako powtórzenia tandemowe. Następująca sekwencja jest przykładem tandemnie powtarzanej sekwencji:
Attata ATTATA ATTATA / /ATTATA
genomy, które zawierają te różne klasy sekwencji, przekształcają się w inny sposób niż genomy z pojedynczymi sekwencjami kopii., Zamiast jednej gładkiej krzywej „Cot” można zobaczyć trzy różne krzywe, z których każda reprezentuje inną klasę powtórzeń. Pierwsze sekwencje reanneal są highlyrepetitive sekwencje ponieważ tak wiele ich kopii istnieje w genomie, i ponieważ mają niską złożoność sekwencji. Druga część genomu do reannealu to środkowe powtarzalne DNA, a końcowa część do reannealu to pojedyncza kopia DNA., Poniższy diagram przedstawia krzywą „Cot” dla „typowego” genomu eukariotycznego
poniższa tabela przedstawia rozkład sekwencji dla wybranych gatunków.
| gatunki | rozkład sekwencji |
|---|---|
| bakterie | 99.,27% średniej powtarzalności 8% średniej powtarzalności |
| kukurydza | 30% pojedynczej kopii 40% średniej powtarzalności 20% średniej powtarzalności |
| pszenica | 10% pojedynczej kopii 83% średniej powtarzalności 4% Średniej powtarzalności |
Arabidopsis | 55% pojedyncza kopia 27% średnio powtarzalna 10% wysoce powtarzalna |
przeplatanie sekwencji
mimo że genomy organizmów wyższych zawierają pojedynczą kopię, średnio powtarzalną i wysoce powtarzalną sekwencję DNA, sekwencje te nie są ułożone podobnie u wszystkich gatunków., Wybitny układ nazywa się krótki okresinterspersja. Układ ten charakteryzuje się powtarzającymi się sekwencjami o długości 100-200 bp przeplatanymi między pojedynczymi sekwencjami o długości 1000-2000bp. Układ ten występuje u zwierząt, grzybów i roślin.
drugim typem układu jest interpersja długookresowa. Charakteryzuje się 5000 bp ciągami powtarzających się sekwencji przeplatanych w regionach 35 000 bp pojedynczej kopii DNA. Drosophila jest przykładem gatunku o tym nietypowym układzie sekwencyjnym., W obu przypadkach sekwencje są zwykle ze średniej klasy powtarzalności. Omówiliśmy, gdzie znajdują się wysoce powtarzalne sekwencje.
kariotyp chromosomu Eukariotycznego
podczas gdy bakterie mają tylko jeden chromosom, gatunki eukariotyczne mają przynajmniej jedną parę chromosomów. Większość ma więcej niż jedną parę. Innym istotnym punktem jest to, że chromosomy eukariotyczne są wykrywane tylko podczas podziału komórek, a nie podczas wszystkich etapów cyklu komórkowego. Są one w swojej największej formie podczas metafazy, gdy przyłączone są chromatydy siostrzane.,Jest to podstawowy etap, w którym wykonuje się analizę cytogenetyczną.
każdy gatunek charakteryzuje się kariotypem. Kariotyp jest opisem liczby chromosomów w normalnej komórce diploidalnej, jak również ich rozkładu wielkości. Na przykład ludzki chromosom ma 23 pary chromosomów, 22 pary somatyczne i jedną parę chromosomów płciowych. Jednym z ważnych aspektów badań genetycznych jest korelacja zmian w kariotypie ze zmianami w fenotypie osobnika.
jednym z ważnych aspektów genetyki jest korelacja zmian w kariotypie ze zmianami w fenotypie., Na przykład, ludzie, którzy mają dodatkowy chromosom 21mamy zespół Downa. Insercje, delecje i zmiany liczby chromosomów mogą być wykryte przez wykwalifikowanego cytogenetyka, ale korelowanie ich ze specyficznymi fenotypami jest trudne.
pierwszym parametrem rozróżniającym przy opracowywaniu kariotypu jest wielkość i liczba chromosomów. Chociaż jest to użyteczne, nie dostarcza zbyt wielu szczegółów, aby rozpocząć rozwój korelacji między strukturą a funkcją (fenotypem)., Aby jeszcze bardziej odróżnić chromosomy, są one traktowane z barwnikiem, który plami DNA w sposób powtarzalny. Po zabarwieniu niektóre regiony są lekko poplamione, a inne są ciężkostopione. Jak opisano powyżej, lekko zabarwione regiony są nazywane euchromatyną, a ciemny zabarwiony region nazywa sięheterochromatyną. Obecnym barwnikiem jest plama Giemsa, a uzyskany wzór nazywa się wzorem g-bandingu.,
paradoks wartości C
oprócz opisu genomu organizmu przez liczbę chromosomów,opisywany jest również przez ilość DNA w komórce haploidalnej. Jest to zwykle wyrażana jako ilość DNA na komórkę haploidalną (zwykle wyrażana jako pikogramy)lub liczba kilobaz na komórkę haploidalną i nazywana jest wartością C. Jedna z cech organizmów eukariotycznych podkreśla specyficzną anomalię, która została wykryta na początku badań molekularnych., Mimo że organizmy eukariotyczne mają 2-10 razy więcej genów niż prokaryoty, mają one o wiele więcej DNA w komórce. Co więcej, ilość DNA na Genom nie koreluje z przypuszczalną złożonością ewolucyjną gatunku.Jest to określane jako paradoks wartości C: ilość DNA w haploidcell organizmu nie jest związana z jego złożonością ewolucyjną. (Innym ważnym punktem, o którym należy pamiętać, jest to, że nie ma związku między liczbą chromosomów a domniemaną złożonością ewolucyjną organizacji.,)
C Values of Organisms Used in Genetic Studies
| Species | Kilobases/haploid genome |
|---|---|
| E. coli |
4.5 x 103 |
| Human |
3.0 x 106 |
| Drosophila |
1.7 x 105 |
| Maize |
2.0 x 106 |
| Aribidopsis |
7.,0 x 104 |
dramatyczny przykład zakresu wartości C można zobaczyć w królestwie roślin, gdzie Arabidopsis reprezentuje niski koniec, a lily (1.0 x 10^8 kb/haploidgenome) wysoki koniec złożoności. W kategoriach wagowych jest to 0,07 pikogramów haploidalnego genomu Arabidopsis i 100 pikogramów na haploidalny lilygenom.
Genom-kompletny zestaw chromosomów odziedziczony po pojedynczym rodzicu;kompletny Składnik DNA jednostki; definicja często wyłącza Genom