Eukaryotische Chromosomenstruktur
Die Länge der DNA im Kern ist weit größer als die Größe des Kompartiments, in dem sie enthalten ist. Um in dieses Abteil zu passen, muss die DNA in irgendeiner Weise untersucht werden. Der Grad, in dem DNA kondensiert ist, wird exprimiertals sein Packungsverhältnis.
Packungsverhältnis-die Länge der DNA geteilt durch die Länge, in dieEs ist verpackt
Zum Beispiel enthält das kürzeste menschliche Chromosom 4,6 x 107 bp DNA (etwa 10 mal die Genomgröße von E. coli)., Dies entspricht 14.000 µm erweiterter DNA. In seinem kondensiertesten Zustand während der Mitose, diechromosom ist etwa 2 µm lang. Dies ergibt ein Packverhältnis von 7000 (14.000/2).
Um das Gesamtpackverhältnis zu erreichen, wird DNA nicht direkt in Finalstruktur von Chromatin verpackt. Stattdessen enthält es mehrere Hierarchien der Organisation.Die erste Ebene der Verpackung wird durch das Wickeln von DNA um einen Proteincore erreicht, um eine „perlenartige“ Struktur zu erzeugen, die als Nukleosom bezeichnet wird. Dies ergibt ein Packungsverhältnis von etwa 6., Diese Struktur ist invariant sowohl in DEREUCHROMATIN und Heterochromatin aller Chromosomen. Die zweite Ebene der Verpackungist das Aufwickeln von Perlen in einer spiralförmigen Struktur, die als 30-nm-Faser bezeichnet wird und sowohl in Interphasenchromatin als auch in mitotischen Chromosomen vorkommt. Dadurch erhöht sich das Packverhältnis auf etwa 40. Die endgültige Verpackung erfolgt, wenn die Faser in Schleifen, Gerüsten und Domänen organisiert ist, die ein Finalpackverhältnis von etwa 1000 in Interphasenchromosomen und etwa 10.000 inmitotischen Chromosomen ergeben.,
Eukaryotische Chromosomen bestehen aus einem DNA-Protein-Komplex, der kompakt organisiert ist, wodurch die große Menge an DNA im Zellkern gespeichert werden kann. Die Untereinheit Bezeichnung des Chromosoms ist Chromatin.Die grundlegende Einheit des chromatins ist das nukleosom.,romatin – die Einheit der Analyse des Chromosoms; Chromatin reflektiertdie allgemeine Struktur des Chromosoms, ist aber nicht einzigartig für irgendein Besondereschromosom
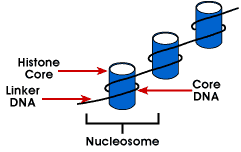
Nukleosom – einfachste Verpackungsstruktur der DNA, die inalle eukaryotischen Chromosomen; DNA ist um einen Oktamer von kleinen basischen gewickeltproteine genannt Histone; 146 bp ist um den Kern gewickelt und die verbliebenenbasen verbinden sich mit dem nächsten Nukleosom; Diese Struktur verursacht negative Supercoiling
Das Nukleosom besteht aus etwa 200 bp um ein histon octamerdas enthält zwei Kopien von Histonproteinen H2A, H2B, H3 und H4., Diese sind als Kernhistone bekannt. Histone sind basische Proteine, die eine Affinität habenfür DNA und sind die am häufigsten vorkommenden Proteine, die mit DNA assoziiert sind. Die Aminosäuresequenz dieser vier Histone ist konserviert, was auf eine ähnliche Funktion hindeutetfür alle.
Die Länge der DNA, die mit der Nukleosomeneinheit assoziiert ist, variiert zwischenspezies. Unabhängig von der Größe sind jedoch zwei DNA-Komponenten beteiligt. CoreDNA ist die DNA, die tatsächlich mit dem Histon Octamer assoziiert ist.Dieser Wert ist invariant und beträgt 146 Basispaare., Die Kern-DNA bildet zwei Schleifen um den Octamer, und dies erlaubt zwei Regionen, die 80 bp auseinander liegen, in die Nähe gebracht zu werden. Somit können zwei Sequenzen, die weit voneinander entfernt sind, mit demselben regulatorischen Protein interagieren, um die Genexpression zu kontrollieren. Die DNA, die zwischen jedem Histon-Oktamer liegt, wird als Linker-DNA bezeichnet und kann in der Länge von 8 bis 114 Basenpaaren variieren. Diese Variation ist spezifisch, aber die Variation der Linker-DNA-Länge wurde auch mit dem Entwicklungsstadium des Organismus oder bestimmten Regionen des Genoms in Verbindung gebracht.,
Die nächste Organisationsebene des Chromatins ist die 30 nm Faser. Dies scheint eine Magnetstruktur mit etwa 6 Nukleosomen pro Umdrehung zu sein. Dies ergibt ein Packungsverhältnis von 40, was bedeutet, dass jeder 1 µm entlang der Axis 40 µm DNA enthält. Die Stabilität dieser Struktur erfordert diepräsenz des letzten Mitglieds der Histongenfamilie, Histon H1., Becauseexperiments, dass der Streifen H1 aus chromatin pflegen das nukleosom, aber nichtder 30-nm-Struktur ergab, dass die H1 ist wichtig für die stabilizationof 30 nm-Struktur.
Die Endstufe der Verpackung ist durch die 700 nm-Struktur gekennzeichnet, die im Metaphasenchromosom zu sehen ist. Das kondensierte Stück Chromatin hat eine acharakteristische Gerüststruktur, die in Metaphasechromosomen nachgewiesen werden kann. Dies scheint das Ergebnis einer ausgedehnten Schleife des DN auf dem Chromosom zu sein.
Die letzten Definitionen, die dargestellt werden müssen, sind Euchromatin Undheterochromatin., Wenn Chromosomen mit Farbstoffen gefärbt sind, scheinen sie zu habenalternative leicht und dunkel gefärbte Regionen. Die leicht gefärbten Regionensind Euchromatin und enthalten Einkopie, genetisch aktive DNA. Die stark gefärbten Regionen sind heterochromatisch und enthalten sich wiederholende Sequenzen, die genetisch inaktiv sind.
Zentromer und Telomere
Zentromer und Telomere sind zwei wesentliche Merkmale aller eukaryotischen Chromosomen. Jede bietet eine einzigartige Funktion, die für die Stabilität des Chromosoms absolut notwendig ist., Zentromer sind für die Segregation des Zentromers während Meiose und Mitose erforderlich, und Teleomere sorgen für terminale Stabilität des Chromosoms und gewährleisten sein Überleben.
Zentromer sind jene kondensierten Regionen innerhalb des Chromosoms, die für die genaue Segregation der replizierten chromosomeduring Mitose und Meiose verantwortlich sind. Wenn Chromosomen gefärbt sind, zeigen sie typischerweise eine dunkel gefärbte Region, die das Zentromer ist. Während der Mitose muss sich das Zentromer, das von den Schwesterchromatiden geteilt wird, teilen, damit die Chromatiden zu Wandernpositpole der Zelle., Auf der anderen Seite, während der ersten meiotischen Teilungdas Zentromer der Schwesterchromatiden muss intakt bleiben, während sie während meiosisII wie während der Mitose wirken müssen. Daher ist das Zentromer eine wichtige Komponente der Chromosomenstruktur und-segregation.
Innerhalb der Zentromerregion haben die meisten Arten mehrere Stellen, an denen sich Spindelfasern anhaften, und diese Stellen bestehen sowohl aus DNA als auch aus Protein. Der tatsächliche Ort, an dem die Anheftung stattfindet, wird Kinetochor genannt und besteht sowohl aus DNA als auch aus Protein. Die DNA-Sequenz innerhalb dieser Regionen wird GENANNTCEN-DNA., Da CEN-DNA von einem Chromosom zum anderen bewegt werden kann und dem Chromosom dennoch die Fähigkeit zur Trennung bietet, dürfen diese Sequenzen keine andere Funktion bieten.
Typischerweise ist CEN-DNA etwa 120 Basenpaare lang und besteht aus mehreren Unterdomänen, CDE-I, CDE-II und CDE-III. Mutationen in den ersten beiden Unterdomänen haben keinen Einfluss auf die Segregation, aber eine Punktmutation in der CDE-III-Unterdomäne eliminiert vollständig die Fähigkeit des Zentromers, während der Chromosomensegregation zu funktionieren. , Daher muss CDE-III aktiv an der Bindung der Spindelfasern an das Zentromer beteiligt sein.
Die Proteinkomponente des Kinetochors wird erst jetzt charakterisiert. Ein Komplex von drei Proteinen namens Cbf-III bindet an normale CDE-III-Regionen, kann jedoch nicht an eine CDE-III-Region mit einer Punktmutation binden, die die mitotische Segregation verhindert. Darüber hinaus eliminieren Mutanten der Gene, die für die Cbf-III-Proteine kodieren, auch die Fähigkeit der Chromosomen, sich während der Mitose zu trennen., Zusätzliche Analysen der DNA – und Proteinkomponenten des Zentromers sind notwendig, um die Mechanik der Chromosomenseigerung vollständig zu verstehen.
Telomere sind die Region der DNA am Ende des linearen eukaryotischen Chromosoms, die für die Replikation und Stabilität des Chromosoms erforderlich sind.McClintock erkannte ihre Besonderheiten, als sie bemerkte, dass, wenn zwei Chromosomen in einer Zelle gebrochen wurden, das Ende eines an das andere anhängen könnteund umgekehrt. Was sie nie beobachtete, war die Anheftung des gebrochenen Chromosoms an das Ende eines ungebrochenen Chromosoms., So sind die Enden von gebrochenen Chromosomensind klebrig, während das normale Ende nicht klebrig ist, was darauf hindeutet, dass die Enden Vonchromosomen haben einzigartige Eigenschaften. Normalerweise, aber nicht immer, ist die telomericDNA heterochromatisch und enthält direkte tandemartig wiederholte Sequenzen. Die nachfolgende Tabelle zeigt die Wiederholungssequenzen mehrerer Arten. Diese haben oft die Form (T/A)xGy wobei x zwischen 1 und 4 liegt und y größer als 1 ist.,
Telomere Repeat Sequences
| Species | Repeat Sequence |
|---|---|
| Arabidopsis | TTTAGGG |
| Human | TTAGGG |
| Oxytricha | TTTTGGGG |
| Slime Mold | TAGGG |
| Tetrahymena | TTGGGG |
| Trypanosome | TAGGG |
| Yeast | (TG)1-3TG2-3 |
Notice that the number of TG sequences and the number of cytosines in the yeast sequence varies., Zumindest für Hefe wurde gezeigt, dass verschiedene Stämme unterschiedliche Teleomerenlängen enthalten und dass die Länge unter genetischer Kontrolle steht.
Die primäre Schwierigkeit bei Telomeren ist die Replikation des verzögerten Strangs.Da für die DNA-Synthese eine RNA-Vorlage (die die freie 3′-Ohgruppe bereitstellt) erforderlich ist, um die DNA-Replikation zu primieren, und diese Vorlage schließlich abgebaut wird, verbleibt am Ende des Chromosoms eine kurze einzelsträngige Region. Diese Region wäre anfällig für Enzyme, die einzelsträngige DNA abbauen., Das Ergebnis wäre, dass die Länge des Chromosoms nach jeder Teilung verkürzt würde. Aber das wird nicht gesehen.
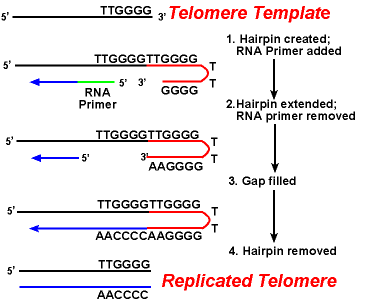
Die Wirkung der Telomerase-Enzyme sorgt dafür, dass die Enden der nachlaufenden Stränge korrekt repliziert werden. Ein gut untersuchtes System umfasst den Tetrahymena-Protozoen-Organismus. Die Telomere dieses Organismus enden in der Sequenz 5′ – TTGGGG-3′. Die Telomerase fügt eine Reihe von 5′-TTGGGG-3′ Wiederholungen an den Enden des verzögerten Strangs hinzu. Eine Haarnadel tritt auf, wenn ungewöhnliche Basenpaare zwischen Guaninresten in der gleichen Form auftreten., Als nächstes wird der RNA-Primer entfernt, und das 5′ – Ende des verzögerten Strangs kann für die DNA-Synthese verwendet werden. Ligation tritt zwischen dem fertigen verzögerten Strang und der Haarnadel auf. Schließlich wird die Haarnadel bei der 5′-TTGGGG-3′ – Wiederholung entfernt. So wird das Ende des Chromosoms getreu repliziert. Die folgende Abbildung zeigt diese Schritte.,
Die Replikation von Telomeren
Analyse von DNA-Sequenzen in eukaryotischen Genomen
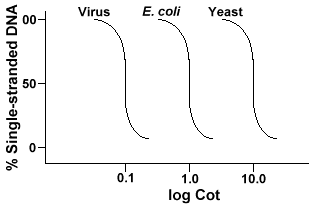
Die Technik, mit der die Sequenzkomplexität eines Genoms bestimmt wirdvolviert die Denaturierung und Renaturierung von DNA. DNA wird durch Erhitzen denaturiert, wodurch die H-Bindungen geschmolzen werden und die DNA einzelsträngig wird. Wenn die DNA schnell abgekühlt ist, bleibt die DNA einzelsträngig. Aber wenn die DNAis langsam abkühlen lassen, Sequenzen, die komplementär sind, werden sich gegenseitig finden und schließlich wieder Basenpaar., Die Geschwindigkeit, mit der die DNA neu reißt(ein anderer Begriff für Renatur), ist eine Funktion der Spezies, aus der die DNAwar isoliert. Unten ist eine Kurve, die aus einem einfachen Genom erhalten wird.
Die Y-Achse ist der Prozentsatz der DNA, der einzelsträngig bleibt. Dies wird als Verhältnis der Konzentration von einzelsträngiger DNA (C)zur Gesamtkonzentration der Start-DNA (Co) exprimiert. Die X-Achse isteine Logskala des Produkts der Anfangskonzentration von DNA (in Mol/Liter)multipliziert mit der Zeitdauer, in der die Reaktion ablief (in Sekunden)., Die Bezeichungfür diesen Wert ist Cot und wird als „Cot“ – Wert bezeichnet. Die Kurve selbstwird als“ Cot “ – Kurve bezeichnet. Wie zu sehen ist, ist die Kurve ziemlich glatt, was darauf hindeutet, dass das Reannealing verlangsamt, aber allmählich über einen Zeitraum von Zeit auftritt. Ein besonderer Wert, der nützlich ist, ist Cot½, der Kot-Wert, bei dem die Hälfte der DNA wieder aufgetaucht ist.
Schritte, die an DNA-Denaturierungs-und Renaturierungsexperimenten beteiligt sind
1. Scheren Sie die DNA auf eine Größe von etwa 400 bp.
2. Denaturieren Sie die DNA durch Erhitzen auf 100oC.
3. Langsam abkühlen und Proben in verschiedenen Zeitintervallen entnehmen.
4., Bestimmen Sie die % einzelsträngige DNA zu jedem Zeitpunkt.
Die Form einer „Cot“ – Kurve für eine bestimmte Art ist eine Funktion von zwei Faktoren:
- die Größe oder Komplexität des Genoms; und
- die Menge an sich wiederholender DNA innerhalb des Genoms
Wenn wir die „Cot“ – Kurven des Genoms von drei Arten wie Bakteriophagelambda, E. coli und Hefe zeichnen,werden wir sehen, dass sie die gleiche Form haben, aber die Cot-Kurve der Hefe wird am größten sein, E. coli nextand lambda kleinste., Je größer die Genomgröße ist, desto länger wird es dauern, bis eine Sequenz auf ihre komplementäre Sequenz in der Lösung stößt. Dies liegt daran, dass zwei komplementäre Sequenzen aufeinander treffen müssen, bevor sie sich paaren können. Je komplexer das Genom ist, das heißt, je mehr einzigartige Sequenzen verfügbar sind, desto länger dauert es, bis sich zwei komplementäre Sequenzen begegnen und paaren. Angesichts ähnlicher Konzentrationen in der Lösung wird es dann eine komplexere Spezies lange dauern, bis sie ihre Größe erreicht hat .,
Wiederholte DNA-Sequenzen, DNA-Sequenzen, die mehr als einmal im Genom der Spezies gefunden werden, haben charakteristische Auswirkungen auf“ Cot “ – Kurven.Wenn eine bestimmte Sequenz zweimal im Genom dargestellt wird, hat sie zwei komplexe Sequenzen zum Paaren und hat als solche einen Cot-Wert, der halb so groß ist wie eine Sequenz, die nur einmal im Genom dargestellt wird.
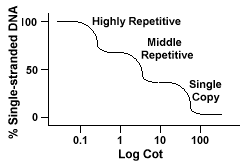
Eukaryotische Genome haben tatsächlich eine breite Palette von Sequenzen, die auf verschiedenen Wiederholungsebenen dargestellt werden., Einzelne Kopiersequenzen finden sich ein paar Mal im Genom. Viele der Sequenzen, die functionalgenes codieren, fallen in diese Klasse. Mitte repetitive DNA werden von 10s -1000 mal im Genom gefunden. Beispiele hierfür wären rRNA und Trnagene und Speicherproteine in Pflanzen wie Mais. Mittlere repetitive DNAcan variieren von 100-300 bp bis 5000 bp und können im gesamten Genom verteilt werden.Die häufigsten Sequenzen finden sich in der sich stark wiederholenden DNAclass., Diese Sequenzen werden von 100.000 bis 1 Million Mal im Genom gefundenund kann in der Größe von einigen bis zu mehreren hundert Basen in der Länge reichen. Thesesequences finden sich in Regionen des Chromosoms wie Hetero -, zentromere und Telomere und neigen dazu, zu angeordnet werden als tandem-Wiederholungen. Thefollowing ist ein Beispiel für eine tandemly wiederholte Sequenz:
ATTATA ATTATA ATTATA // ATTATA
die Genome enthalten diese verschiedenen Klassen von Sequenzen reanneal in einem differentmanner als genomen nur single copy-Sequenzen., Anstatt eine einfache glatte “ Cot “ – Kurve zu haben, sind drei verschiedene Kurven zu sehen, die jeweils eine andere Wiederholungsklasse darstellen. Die ersten zu reannealisierenden Sequenzen sind die hochrepetitiven Sequenzen, weil so viele Kopien von ihnen im Genom existieren und weil sie eine geringe Sequenzkomplexität aufweisen. Der zweite Teil des Genoms zu reanneal ist die mittlere repetitive DNA, und der letzte Teil toreanneal ist die Einzelkopie DNA., Das folgende Diagramm zeigt die“Cot“ – Kurve für ein“ typisches „eukaryotisches Genom
Die folgende Tabelle gibt die Sequenzverteilung für ausgewählte Arten an.
| Spezies | Sequenzverteilung |
|---|---|
| Bakterien | 99.,27% Mittel Repetitiv 8% Hoch Repetitiv |
| Mais | 30% Einzelkopie 40% Mittel Repetitiv 20% Hoch repetitiv |
| Weizen | 10% Einzelkopie 83% Mittel Repetitiv 4% Hoch repetitiv |
| Arabidopsis | 55% Einzelkopie 27% Middle Repetitive 10% Highly Repetitive |
Sequence Interspersion
Obwohl die Genome höherer Organismen single copy, middlerepetitive und highly repetitive DNA sequences enthalten, sind diese Sequenzen nicht in allen Spezies gleich angeordnet., Die prominente Anordnung wird kurze Periodinterspersion genannt. Diese Anordnung ist durch wiederholte Sequenzen gekennzeichnet100-200 bp in der Länge zwischen einzelnen Kopiersequenzen, die 1000-2000bp lang sind. Diese Anordnung findet sich in Tieren, Pilzen und Pflanzen.
Die zweite Art der Anordnung ist langperiodige Interspersion. Dies ist gekennzeichnet durch 5000 bp-Strecken wiederholter Sequenzen, die in Regionen von 35.000 bp Einzelkopie-DNA durchsetzt sind. Drosophila ist ein Beispiel einer Spezies mit dieser ungewöhnlichen Sequenzanordnung., In beiden Fällen gibt es in der Regel aus der Mittelklasse. Wir diskutierten darüber, wo sich stark wiederholende Sequenzen befinden.
Eukaryotischer Chromosomenkaryotyp
Während Bakterien nur ein einziges Chromosom haben, haben eukaryotische Spezies mindestens ein Chromosomenpaar. Die meisten haben mehr als ein paar. Ein weiterer relevanter Punkt ist, dass eukaryotische Chromosomen nur während der Zellteilung und nicht in allen Stadien des Zellzyklus nachgewiesen werden. Sie sind in ihrer am meisten verdichteten Form während der Metaphase, wenn die Schwesterchromatiden befestigt sind.,Dies ist das primäre Stadium, in dem eine zytogenetische Analyse durchgeführt wird.
Jede Art ist durch einen Karyotyp gekennzeichnet. Der Karyotyp ist eine Beschreibung der Anzahl der Chromosomen in der normalen diploiden Zelle sowie ihrer Größenverteilung. Zum Beispiel hat das menschliche Chromosom 23 Paareder Chromosom, 22 somatische Paare und ein Paar Geschlechtschromosomen. Ein wichtiger Aspekt der genetischen Forschung ist die Korrelation von Veränderungen des Karyotyps mit Veränderungen im Phänotyp des Individuums.
Ein wichtiger Aspekt der Genetik ist die Korrelation von Veränderungen im Karyotyp mitVeränderungen im Phänotyp., Zum Beispiel Menschen, die ein zusätzliches Chromosom 21 habenhaben Down-Syndrom. Insertionen, Deletionen und Veränderungen der Chromosomenzahlkann vom erfahrenen Zytogenetiker nachgewiesen werden, aber es ist schwierig, diese mit spezifischen Phänotypen zu korrelieren.
Der erste diskriminierende Parameter bei der Entwicklung eines Karyotyps ist die Größe und Anzahl der Chromosomen. Obwohl dies nützlich ist, bietet es nichtgenug Details, um die Entwicklung einer Korrelation zwischen Struktur und Funktion (Phänotyp) zu beginnen., Zur weiteren Unterscheidung zwischen Chromosomen, siewerden mit einem Farbstoff behandelt, der die DNA reproduzierbar färbt. Nach dem Färben sind einige der Regionen leicht fleckig und andere sind schwergefärbt. Wie oben beschrieben, werden die leicht gefärbten Regionen genannteuchromatin, und die dunkel gefärbte Region wird genanntheterochromatin. Der aktuelle Farbstoff der Farbe ist der Giemsa-Fleck, und das resultierende Muster wird als G-Banding-Muster bezeichnet.,
C-Wert-Paradoxon
Neben der Beschreibung des Genoms eines Organismus anhand seiner Chromosomenzahl wird es auch anhand der DNA-Menge in einer haploiden Zelle beschrieben. Dies wird normalerweise als die Menge an DNA pro haploide Zelle (normalerweise als Picogramme exprimiert)oder die Anzahl der Kilobasen pro haploide Zelle ausgedrückt und wird als C-Wert bezeichnet. Ein unmittelbares Merkmal von eukaryotischen Organismen hebt eine spezifische Anomalie hervor, die früh in der molekularen Forschung entdeckt wurde., Obwohl eukaryotische Organismen erscheinen, um 2-10 mal so viele Gene wie Prokaryoten zu haben, haben sie viele Größenordnungen mehr DNA in der Zelle. Darüber hinaus korreliert die Menge an DNA pro Genom nicht mit der vermuteten evolutionären Komplexität einer Spezies.Dies wird als C-Wert-Paradoxon angegeben: Die Menge an DNA in der Haploidzelle eines Organismus hängt nicht mit seiner evolutionären Komplexität zusammen. (Another Important point to keep in mind is that there is no relationship between the number of chromosomes and the presumed evolutionary complexity of an organism.,)
C Values of Organisms Used in Genetic Studies
| Species | Kilobases/haploid genome |
|---|---|
| E. coli |
4.5 x 103 |
| Human |
3.0 x 106 |
| Drosophila |
1.7 x 105 |
| Maize |
2.0 x 106 |
| Aribidopsis |
7.,0 x 104 |
Ein dramatisches Beispiel für den Bereich der C-Werte ist im Pflanzenkönigreich zu sehen, in dem Arabidopsis das untere Ende darstellt und lily (1.0 x 10^8 kb/haploidgenom) das hohe Ende der Komplexität. Gewichtsmäßig sind dies 0,07 Picogramme pro haploid Arabidopsis Genom und 100 Picogramme pro haploid Lilygenom.
Genom-der vollständige Chromosomensatz, der von einem einzelnen Elternteil geerbt wurde;die vollständige DNA-Komponente eines Individuums; Die Definition schließt Oftorganellen aus