structure des chromosomes eucaryotes
La longueur de l’ADN dans le noyau est bien supérieure à la taille du compartiment dans lequel il est contenu. Pour entrer dans ce compartiment, l’ADN doit êtrecondensé d’une manière ou d’une autre. Le degré auquel L’ADN est condensé est exprimécomme son rapport d’emballage.
rapport D’emballage – la longueur de L’ADN divisée par la longueur dans laquelleil est emballé
Par exemple, le chromosome humain le plus court contient 4,6 x 107 PB d’ADN(environ 10 fois la taille du génome d’E. coli)., Cela équivaut à 14 000 µm d’ADN étendu. Dans son état le plus condensé pendant la mitose, lechromosome mesure environ 2 µm de long. Cela donne un rapport d’emballage de 7000 (14 000/2).
pour atteindre le rapport d’emballage global, L’ADN n’est pas emballé directement dans la structure finale de la chromatine. Au lieu de cela, il contient plusieurs hiérarchies d’organisation.Le premier niveau d’emballage est obtenu par l’enroulement de L’ADN autour d’un proteincore pour produire une structure « en forme de perle » appelée nucléosome. Cela donne un rapport d’emballage d’environ 6., Cette structure est invariante dans leeuchromatine et l’hétérochromatine de tous les chromosomes. Le deuxième niveau d’emballage est l’enroulement de perles dans une structure hélicoïdale appelée fibre de 30 nm qui se trouve à la fois dans la chromatine interphasique et les chromosomes mitotiques. Cette structure augmente le rapport d’emballage à environ 40. L’emballage final se produit lorsque la fibre est organisée en boucles, échafaudages et domaines qui donnent un rapport d’emballage final d’environ 1000 dans les chromosomes interphasiques et d’environ 10 000 chromosomes inmitotiques.,
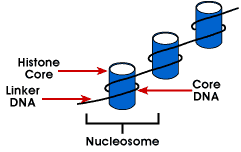
Les chromosomes eucaryotes sont constitués d’un complexe ADN-protéine qui est organisé de manière compacte, ce qui permet de stocker une grande quantité d’ADN dans le noyau de la cellule. La désignation de sous-unité du chromosome est la chromatine.L’unité fondamentale de la chromatine est le nucléosome.,romatin – l’unité d’analyse du chromosome; la chromatine reflète la structure générale du chromosome mais n’est unique à aucun particularchromosome
nucléosome-structure d’emballage la plus simple de L’ADN qui se trouve dans tous les chromosomes eucaryotes; L’ADN est enroulé autour d’un octamère de petites protéines de base appelées histones; 146 bp est enroulé autour 200 bp enroulé autour d’un octamère histone qui contient deux copies des protéines histones H2A, H2B, H3 et H4., Ceux-ci sont connus comme les histones de base. Les Histones sont des protéines de base qui ont une affinité pour L’ADN et sont les protéines les plus abondantes associées à L’ADN. La séquence aminoacide de ces quatre histones est conservée suggérant une fonction similaire pour tous.
La longueur de L’ADN associée à l’unité nucléosome varie entreespèces. Mais quelle que soit la taille, deux composants de l’ADN sont impliqués. CoreDNA est l’ADN qui est réellement associé à l’octamère d’histone.Cette valeur est invariante et est de 146 paires de bases., L’ADN de noyau forme deux boucles autour de l’octamère, et ceci permet deux régions qui sont 80 bp à part Tobe mis dans la proximité étroite. Ainsi, deux séquences éloignées peuventinteract avec la même protéine régulatrice pour contrôler l’expression des gènes. L’ADN qui se trouve entre chaque octamère d’histone est appelé l’éditeur de liens Dnaet peut varier en longueur de 8 à 114 paires de bases. Cette variation est spécifique, mais la variation de la longueur de l’ADN du Lieur a également été associée au stade de développement de l’organisme ou à des régions spécifiques du génome.,
Le prochain niveau de l’organisation de la chromatine est le 30 nm de la fibre. Cela semble être une structure de solénoïde avec environ 6 nucléosomes par tour. Ceci donne un rapport d’emballage de 40, ce qui signifie que tous les 1 µm le long de l’axiscontient 40 µm d’ADN. La stabilité de cette structure nécessite leprésence du dernier membre de la famille des gènes de l’histone, l’histone H1., Parce que les expériences qui enlèvent H1 de la chromatine maintiennent le nucléosome, mais pas la structure de 30 nm, il a été conclu que H1 est important pour la stabilisation de la structure de 30 nm.
le niveau final de conditionnement est caractérisé par la structure de 700 nm observée dans le chromosome de la métaphase. Le morceau condensé de chromatine a une structure d’échafaudage caractéristique qui peut être détectée dans les métaphasechromosomes. Cela semble être le résultat d’une boucle étendue de la DNAin le chromosome.
Les dernières définitions qui doivent être présentées sont euchromatine ethétérochromatine., Lorsque les chromosomes sont colorés avec des colorants, ils semblent avoirrégions légèrement et sombres en alternance. Les régions légèrement tachéessont euchromatine et contiennent une seule copie, ADN génétiquement actif. Les régions faiblement colorées sont hétérochromatines et contiennent des séquences répétitives qui sont génétiquement inactives.
centromères et télomères
Les centromères et les télomères sont deux caractéristiques essentielles de tous les chromosomes eucaryotes. Chacun fournit une fonction unique qui est absolument nécessaire à la stabilité du chromosome., Les centromères sont nécessaires pour la ségrégation du centromère pendant la méiose et la mitose, et les téléomères fournissent une stabilité terminale au chromosome et assurent sa survie.
Les centromères sont les régions condensées du chromosome qui sont responsables de la ségrégation précise des chromosomes répliqués pendant la mitose et la méiose. Lorsque les chromosomes sont colorés, ils montrent généralement une région tachée de noir qui est le centromère. Au cours de la mitose, le centromère partagé par les chromatides sœurs doit se diviser afin que les chromatides puissent migrer vers les pôles de la cellule., D’autre part, pendant la première division méiotiquele centromère des chromatides sœurs doit rester intact, alors que pendant meiosisII ils doivent agir comme ils le font pendant la mitose. Par conséquent, le centromère est un composant important de la structure et de la ségrégation des chromosomes.
dans la région du centromère, la plupart des espèces ont plusieurs endroits où les fibres de fuseau se fixent, et ces sites sont constitués d’ADN ainsi que de protéines. L’emplacement réel où l’attachement se produit s’appelle le kinétochore et est composé à la fois d’ADN et de protéine. La séquence D’ADN dans ces régions est appelée ADN cen., Étant donné que L’ADN CEN peut être déplacé d’un chromosome à un autre tout en offrant au chromosome la capacité de se séparer, ces séquences ne doivent pas fournir d’autre fonction.
typiquement, L’ADN CEN mesure environ 120 paires de bases et se compose de plusieurs sous-domaines, CDE-I, CDE-II et CDE-III. Les Mutations dans les deux premiers sous-domaines n’ont aucun effet sur la ségrégation, mais une mutation ponctuelle dans le sous-domaine CDE-III élimine complètement la capacité du centromère à fonctionner pendant la ségrégation chromosomique., CDE-III doit donc être activement impliqué dans la liaison des fibres de la broche au centromère.
la composante protéique du kinétochore n’est que maintenant en cours de caractérisation. Un complexe de trois protéines appelé Cbf-III se lie aux régions CDE-III normales mais ne peut pas se lier à une région CDE-III avec une mutation ponctuelle qui empêche la ségrégation mitotique. De plus, les mutants des gènes codant pour les protéines Cbf-III éliminent également la capacité des chromosomes à se séparer pendant la mitose., Des analyses supplémentaires de l’ADN et des composants protéiques du centromère sont nécessaires pour bien comprendre la mécanique de la ségrégation chromosomique.
les télomères sont la région de L’ADN située à l’extrémité du chromosome eucaryote linéaire qui est nécessaire à la réplication et à la stabilité du chromosome.McClintock a reconnu leurs particularités quand elle a remarqué, que si deuxchromosomes étaient cassés dans une cellule, l’extrémité de l’un pourrait s’attacher à l’autreet vice versa. Ce qu’elle n’a jamais observé, c’est l’attachement de l’extrémité cassée à l’extrémité d’un chromosome ininterrompu., Ainsi, les extrémités des chromosomes cassés sont collantes, alors que l’extrémité normale n’est pas collante, ce qui suggère que les extrémités des chromosomes ont des caractéristiques uniques. Habituellement, mais pas toujours, l’ADNM télomérique est hétérochromatique et contient des séquences directes répétées en tandem. Le tableau suivant montre les séquences répétées de plusieurs espèces. Ceux-ci sont souvent de la forme (T/A)xGy où x est compris entre 1 et 4 et y est supérieur à 1.,
Telomere Repeat Sequences
| Species | Repeat Sequence |
|---|---|
| Arabidopsis | TTTAGGG |
| Human | TTAGGG |
| Oxytricha | TTTTGGGG |
| Slime Mold | TAGGG |
| Tetrahymena | TTGGGG |
| Trypanosome | TAGGG |
| Yeast | (TG)1-3TG2-3 |
Notice that the number of TG sequences and the number of cytosines in the yeast sequence varies., Au moins pour la levure, il a été démontré que différentes souches contiennent différentes longueurs de téléomères et que la longueur est sous contrôle génétique.
La principale difficulté avec des télomères est la réplication du brin en retard.Puisque la synthèse D’ADN exige un modèle D’ARN (qui fournit le 3′-OHgroup libre) pour amorcer la réplication D’ADN, et ce modèle est finalement dégradé, une région simple brin courte serait laissée à l’extrémité du chromosome. Cette région serait sensible aux enzymes qui dégradent L’ADN monocaténaire., Le résultat serait que la longueur du chromosome serait raccourcie après chaque division. Mais ce n’est pas vu.
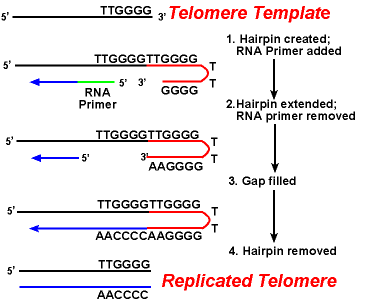
L’action des enzymes télomérase garantit que les extrémités des brins retardants sont répliquées correctement. Un système bien étudié implique L’organisme protozoaire Tetrahymena. Les télomères de cet organisme se terminent par la séquence 5 ‘- TTGGGG-3′. La télomérase ajoute une série de répétitions 5′-TTGGGG-3′ aux extrémités du brin en retard. Une épingle à cheveux se produit lorsque des paires de bases inhabituelles entre les résidus de guanine sous la forme répétée., Ensuite, l’amorce D’ARN est retirée et l’extrémité 5′ du brin en retard peut être utilisée pour la synthèse de l’ADN. La ligature se produit entre le brin de retard fini et l’épingle à cheveux. Enfin, l’épingle à cheveux est retirée à la répétition 5′-TTGGGG-3’. Ainsi, l’extrémité du chromosome est fidèlement reproduite. La figure suivante montre ces étapes.,
la réplication des télomères
analyse des séquences D’ADN dans les génomes eucaryotes
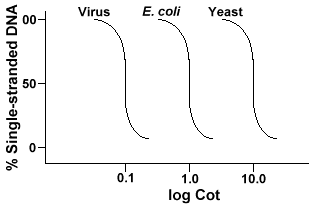
la technique utilisée pour déterminer la complexité de séquence de tout génomeinvolve la dénaturation et renaturation de l’ADN. L’ADN est dénaturedby chauffant qui fait fondre les liaisons H et rend L’ADN simple brin. Si L’ADN est rapidement refroidi, l’ADN reste monocaténaire. Mais si les Dnis ont laissé refroidir lentement, les séquences qui sont complémentaires se retrouveront et éventuellement paire de base à nouveau., La vitesse à laquelle l’ADN réannèle(un autre terme pour renature) est fonction de l’espèce à partir de laquelle L’ADN a été isolé. Voici une courbe obtenue à partir d’un génome simple.
l’axe des Y est le pourcentage de L’ADN qui reste simple brin. Ceci est exprimé comme un rapport de la concentration d’ADN simple brin (C)à la concentration totale de L’ADN de départ (Co). L’axe X estune échelle logarithmique du produit de la concentration initiale d’ADN (en moles/litre)multipliée par la durée de la réaction (en secondes)., La désignation pour cette valeur est Cot et est appelée la valeur « Cot ». La courbe elle-mêmeest appelé une courbe « lit ». Comme on peut le voir, la courbe est plutôt lisse, ce qui indique que le reannealing se produit en ralentissant mais progressivement sur une période de temps. Une valeur particulière qui est utile est Cot½, valeur de theCot où la moitié de l’ADN a recuit.
étapes impliquées dans les expériences de dénaturation et de Renaturation de l’ADN
1. Cisaillez l’ADN à une taille d’environ 400 PB.
2. Dénaturer L’ADN en chauffant à 100oC.
3. Refroidir lentement et prélever des échantillons à différents intervalles de temps.
4., Déterminez le % D’ADN monocaténaire à chaque point temporel.
la forme d’une courbe « Cot » pour une espèce donnée est fonction de deux facteurs:
- La taille ou la complexité du génome; et
- la quantité d’ADN répétitif dans le génome
Si nous traçons les courbes « Cot » du génome de trois espèces telles que bacteriophagelambda, E. coli et la levure,nous verrons qu’elles ont la même forme, mais le Cot½ de la levure sera plus grand, E. coli suivant et lambda le plus petit., Physiquement, plus la taille du génome est grande, plus il faudra longtemps pour qu’une séquence rencontre sa séquence complémentaire dans la solution. C’est parce que deux séquences complémentaires doivent rencontrereach autre avant qu’ils puissent apparier. Plus le génome est complexe, c’est-à-dire plus de séquences uniques disponibles, plus il faudra de temps pour que deux séquences complémentaires se rencontrent et se couplent. Compte tenu de concentrations similaires en solution, il faudra alors à une espèce plus complexe plus longtemps pour atteindre le Cot½ .,
séquences d’ADN Répétées, des séquences d’ADN que l’on retrouve plus d’une fois dans le génome de l’espèce, ont des effets sur le « Berceau » des courbes.Si une séquence spécifique est représentée deux fois dans le génome, elle aura deux séquences complémentaires à apparier et, en tant que telle, aura une valeur Cot halfas grande comme une séquence représentée une seule fois dans le génome.
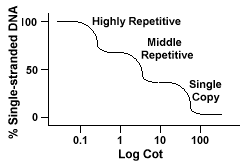
les génomes eucaryotes ont en fait un large éventail de séquences qui sont représentées à différents niveaux de répétition., Les séquences d’une seule copie sont trouvées une foisou quelques fois dans le génome. La plupart des séquences qui encodent des fonctionnalitésles gènes entrent dans cette classe. L’ADN répétitif moyen se trouve de 10s à 1000 fois dans le génome. Les ARNr, les trnagènes et les protéines de stockage dans des plantes comme le maïs en sont des exemples. Dnac répétitif moyen peut varier de 100-300 bp à 5000 bp et peut être dispersé dans tout le génome.Les séquences les plus abondantes se trouvent dans la Dnaclasse très répétitive., Ces séquences se trouvent de 100 000 à 1 million de fois dans le génomeet peuvent varier en taille de quelques à plusieurs centaines de bases de longueur. Ces séquences se trouvent dans des régions du chromosome telles que l’hétérochromatine,les centromères et les télomères et ont tendance à être disposées en tandem. Le suivant est un exemple d’une séquence répétée en tandem:
ATTATA ATTATA ATTATA / /ATTATA
génomes qui contiennent ces différentes classes de séquences reanneal dans un differentmanner que les génomes avec seulement des séquences de copie unique., Au lieu d’avoir une seule courbe « Cot » lisse, trois courbes distinctes peuvent être vues, chacune représentant une classe de répétition différente. Les premières séquences à réanneler sont les séquences très répétitives parce qu’il en existe tant de copies dans le génome et parce qu’elles ont une faible complexité de séquence. La deuxième partie de thegenome à reanneal est L’ADN répétitif moyen, et la dernière partie toreanneal est L’ADN à copie unique., Le diagramme suivant représente le « Berceau »de la courbe pour un « typique » du génome eucaryote
Le tableau suivant donne la séquence de distribution pour les espèces sélectionnées.
| Espèces, | Séquence de Distribution |
|---|---|
| Bactéries | 99.,27% moyen répétitif 8% très répétitif |
| maïs | 30% exemplaire unique 40% moyen répétitif 20% très répétitif |
| blé | 10% exemplaire unique 83% moyen répétitif 4% très répétitif |
| Arabidopsis | 55% exemplaire unique 27% moyen répétitif 10% très répétitif |
séquence interspersion
même si les génomes des organismes supérieurs contiennent des séquences D’ADN à copie unique, moyennement compétitives et très répétitives, ces séquences ne sont pas arrangées de manière similaire chez toutes les espèces., L’arrangement proéminent est appelé court periodinterspersion. Cet arrangement est caractérisé par des séquences répétées100-200 bp de longueur intercalées entre des séquences de copie unique qui sont 1000-2000bp de longueur. Cet arrangement se trouve chez les animaux, les champignons et les plantes.
le deuxième type d’arrangement est l’interspersion à longue période. Ceci est caractérisé par des étirements de 5000 pb de séquences répétées entrecoupées dans des régions de 35 000 PB d’ADN à copie unique. Drosophila est un exemple d’une espèce avec cet arrangement de séquence peu commun., Dans les deux cas, les séquences répétées sont généralement de la classe moyenne répétitive. Nous avons discuté ci-dessus où se trouvent des séquences très répétitives.
caryotype chromosomique eucaryote
alors que les bactéries n’ont qu’un seul chromosome, les espèces eucaryotes ont au moins une paire de chromosomes. La plupart ont plus d’une paire. Un autre point pertinent est que les chromosomes eucaryotes sont détectés ne se produisent que pendant la division cellulaire et non pendant toutes les étapes du cycle cellulaire. Ils sont dans leur forme mostcondensed pendant la métaphase quand les chromatides sœurs sont attachées.,C’est l’étape primaire lors de l’analyse cytogénétique.
Chaque espèce est caractérisée par un caryotype. Le caryotype est une description du nombre de chromosomes dans la cellule diploïde normale, ainsi que de leur distribution de taille. Par exemple, le chromosome humain a 23 pairesde chromosome, 22 paires somatiques et une paire de chromosomes sexuels. Un aspect important de la recherche génétique est de corréler les changements dans le caryotype avec les changements dans le phénotype de l’individu.
un aspect important de la génétique est de corréler les changements de caryotype avec les changements de phénotype., Par exemple, les humains qui ont un chromosome supplémentaire 21avoir le syndrome de Down. Les Insertions, les délétions et les changements dans le nombre de chromosomes peuvent être détectés par le cytogénéticien qualifié, mais il est difficile de les corréler avec des phénotypes spécifiques.
le premier paramètre discriminant lors du développement d’un caryotype est la tailleet le nombre des chromosomes. Bien que cela soit utile, il ne fournit pas assez de détails pour commencer le développement d’une corrélation entre la structure et la fonction (phénotype)., Pour distinguer davantage les chromosomes, ilssont traités avec un colorant qui colore l’ADN de manière reproductible. Après la coloration, certaines régions sont légèrement colorées et d’autres sont fortement colorées. Comme décrit ci-dessus, les régions légèrement colorées sont appeléeseuchromatine, et la région colorée sombre est appeléehétérochromatine. Le colorant actuel de choisi est la teinture Giemsa, et le motif résultant est appelé le motif G-banding.,
C-Valeur Paradoxe
En plus de décrire le génome d’un organisme par le nombre de ses chromosomes,il est aussi décrit par la quantité d’ADN dans une cellule haploïde. Ceci est généralementexprimé comme la quantité d’ADN par cellule haploïde (généralement exprimée en picogrammes)ou le nombre de kilobases par cellule haploïde et est appelé la valeur C. Une caractéristique immédiate des organismes eucaryotes met en évidence une anomalie spécifique qui a été détectée tôt dans la recherche moléculaire., Même si les organismes eucaryotes semblent avoir 2 à 10 fois plus de gènes que les procaryotes, ils ont de nombreux ordres de grandeur plus D’ADN dans la cellule. De plus, la quantité d’ADN par génomeest corrélée non pas à la complexité évolutive présumée d’une espèce.Ceci est indiqué comme le paradoxe de la valeur C: la quantité d’ADN dans l’HAPLOÏDECELLULE d’un organisme n’est pas liée à sa complexité évolutive. (Un autre point important à garder à l’esprit est qu’il n’y a pas de relation entre le nombre de chromosomes et la complexité évolutive présumée d’un organisme.,)
C Values of Organisms Used in Genetic Studies
| Species | Kilobases/haploid genome |
|---|---|
| E. coli |
4.5 x 103 |
| Human |
3.0 x 106 |
| Drosophila |
1.7 x 105 |
| Maize |
2.0 x 106 |
| Aribidopsis |
7.,0 x 104 |
Un exemple spectaculaire de la plage de valeurs C peut être vu dans le royaume végétal où Arabidopsis représente le bas de gamme et lily (1.0 x 10^8 kb/haploïdgénome) le haut de gamme de la complexité. En termes de poids, c’est 0,07 picogramsper génome haploïde Arabidopsis et 100 picogrammes par lilygénome haploïde.
génome – l’ensemble complet des chromosomes hérités d’un seul parent;la composante complète de L’ADN d’un individu; la définition exclut souventorganelles