estrutura do cromossoma eucariótico
o comprimento do ADN no núcleo é muito maior do que o tamanho da compartimentina que está contida. Para caber neste compartimento, o ADN tem de ser cedido de alguma forma. O grau de condensação do ADN é expresso como a sua relação de embalagem.razão de embalagem
– o comprimento do ADN dividido pelo comprimento em que é embalado
por exemplo, o cromossoma humano mais curto contém 4, 6 x 107 bp de ADN(cerca de 10 vezes o tamanho do genoma da E. coli)., Isto é equivalente a 14.000 µm de DNA estendido. Em seu estado mais condensado durante a mitose, o cromossoma tem cerca de 2 µm de comprimento. Isto dá uma razão de embalagem de 7000 (14.000/2).
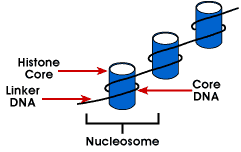
para atingir a razão global de embalagem, o ADN não é embalado directamente na estrutura final da cromatina. Em vez disso, contém várias hierarquias de organização.O primeiro nível de empacotamento é alcançado pelo enrolamento do DNA em torno de um proteincore para produzir uma estrutura “semelhante a contas” chamada de nucleossomo. Isto dá uma proporção de embalagem de cerca de 6., Esta estrutura é invariante tanto na teauchromatina como na heterocromatina de todos os cromossomas. O segundo nível de empacotamento é a colagem de contas em uma estrutura helicoidal chamada de 30 nm fiber que é encontrada em ambos os cromossomos cromatina e mitótico da interfase. Esta estrutura aumenta a relação de embalagem para cerca de 40. A embalagem final ocorre quando a fibra é organizada em loops, andaimes e domínios que dão uma razão de finalpacking de cerca de 1000 nos cromossomas interfase e cerca de 10 000 cromossomas inmitóticos.,os cromossomas eucarióticos consistem num complexo proteico de ADN organizado de forma compacta, que permite armazenar grande quantidade de ADN no núcleo da célula. A subunidade do cromossomo é a cromatina.A unidade fundamental da cromatina é o nucleossomo.,romatin – a unidade de análise do cromossomo; cromatina reflectsthe estrutura geral do cromossomo, mas não é exclusivo para qualquer particularchromosome
Nucleosome – o mais simples estrutura de empacotamento do DNA que é encontrado inall eucarióticas cromossomos; o DNA é enrolado em torno de um octamer de pequeno basicproteins chamado de histonas; 146 bp é enrolada em torno do núcleo e o remainingbases link para a próxima nucleosome; esta estrutura faz com que negativo supercoiling
O nucleosome é composta de cerca de 200 bp enrolado em torno de uma histona octamerthat contém duas cópias de histona proteínas H2A, H2B, H3 e H4., Estes são conhecidos como os histones centrais. Histonas são proteínas básicas que têm uma afinidade para o ADN e são as proteínas mais abundantes associadas ao ADN. A sequência aminoacídica destes quatro histonas é conservada sugerindo uma função similar para todos.
O comprimento do ADN que está associado à unidade nucleossómica varia entre espécies. Mas, independentemente do tamanho, estão envolvidos dois componentes de ADN. CoreDNA é o DNA que está realmente associado com o octâmero de histona.Este valor é invariante e é 146 pares base., O DNA do núcleo forma dois loopsaround the octamer,e isso permite que duas regiões com 80 bp de distância sejam trazidas para a proximidade. Assim, duas sequências que estão muito distantes podem interagir com a mesma proteína reguladora para controlar a expressão do gene. TheDNA que está entre cada octâmero histônico é chamado de linker DNAand pode variar em comprimento de 8 a 114 pares de base. Esta variação é específica, mas a variação no comprimento do ADN linker também tem sido associada à fase de desenvolvimento do organismo ou regiões específicas do genoma.,
O próximo nível de organização da cromatina é o de 30 nm de fibra. Isto parece ser uma estrutura solenóide com cerca de 6 nucleossomas por volta. Isto gera uma razão de embalagem de 40, o que significa que cada 1 µm ao longo da axisconta 40 µm de DNA. A estabilidade desta estrutura requer a presença do último membro da família do gene histone, histone H1., Como os experimentos que extraem H1 da cromatina mantêm o nucleossomo, mas não a estrutura de 30 nm, concluiu-se que H1 é importante para a estabilização da estrutura de 30 nm.
o nível final da embalagem é caracterizado pela estrutura de 700 nm seenina no cromossoma metafase. A peça condensada de cromatina tem estrutura de andaimes acharacterísticos que podem ser detectados em metafasecromossomas. Este parece ser o resultado de um extenso looping do DNAin do cromossomo.
As últimas definições que precisam ser apresentadas são eucromatina eterocromatina., Quando os cromossomas são manchados com corantes, eles parecem ter manchado regiões levemente e escuras. As regiões pouco manchadas são a eucromatina e contêm ADN geneticamente activo de uma única cópia. As regiões manchadas de tinta clara são heterocromatinas e contêm sequências repetitivas que são geneticamente inativas.Centrómeros e telómeros são duas características essenciais de todos os cromossomas eucarióticos. Cada um fornece uma função única que é absolutamente necessária para a estabilidade do cromossomo., Os centrómeros são necessários para a segregação do centrômero durante a meiose e mitose, e os teleômeros proporcionam estabilidade terminal ao cromossomo e garantem a sua sobrevivência.Centrómeros
são as regiões condensadas dentro do cromossoma que são responsáveis pela segregação precisa da mitose e meiose cromossómicas replicadas. Quando os cromossomas são manchados eles tipicamente mostram uma região escura que é o centrômero. Durante a mitose, o centrômero que é compartilhado pelos cromatídeos irmãos deve se dividir para que os cromatídeos possam migrar polos de toopposite da célula., Por outro lado, durante a primeira divisão meiótica, o centrômero dos cromatídeos irmãos deve permanecer intacto, enquanto que durante meiosisII eles devem agir como fazem durante a mitose. Por conseguinte, o centromere é um componente importante da estrutura e da segregação cromossómicas.
dentro da região de centromere, a maioria das espécies tem vários locais onde as fibras de fuso se ligam, e estes locais consistem de DNA, bem como proteínas. A localização real onde o apego ocorre é chamado de cinetócoro e é composto de DNA e proteína. A sequência de ADN nestas regiões chama-se ADN cledcen., Como o DNA CEN pode ser movido de um cromossomo para outro e ainda fornecer ao cromossomo a capacidade de segregar, essas sequências não devem fornecer qualquer outra função.as mutações nos dois primeiros sub-domínios não têm efeito sobre a segregação, mas uma mutação pontual no sub-domínio CDE-III elimina completamente a capacidade do centromere funcionar durante a segregação cromossómica., Portanto, o CDE-III deve estar ativamente envolvido na ligação das fibras do fuso ao centrômero.
O componente proteico do kinetocore só está sendo caracterizado agora. Um complexo de três proteínas chamado Cbf-III liga-se a regiões CDE-III normais, mas não pode se ligar a uma região CDE-III com uma mutação pontual que impede a segregação mitótica. Além disso, os mutantes dos genes que codificam as proteínas Cbf-III também eliminam a capacidade de segregação dos cromossomas durante a mitose., São necessárias análises adicionais dos componentes de ADN e proteínas do centrômero para compreender plenamente a mecânica da segregação cromossómica.telómeros
são a região do ADN no final do eucarioticromossoma linear que são necessários para a replicação e estabilidade do cromossoma.McClintock reconheceu suas características especiais quando ela notou, que se dois cromossomas foram quebrados em uma célula, o fim de um poderia se anexar ao outro e vice-versa. O que ela nunca observou foi o apego do brokenend ao fim de um cromossomo ininterrupto., Assim, as extremidades dos cromossomas quebrados são pegajosas, enquanto que a extremidade normal não é pegajosa, sugerindo que as extremidades dos cromossomas têm características únicas. Normalmente, mas nem sempre, a telomericDNA é heterocromática e contém sequências repetidas em tandemly diretas. O quadro seguinte apresenta as sequências repetidas de várias espécies. Estes são frequentemente Da Forma (T/a)xGy onde x está entre 1 e 4 e y é maior que 1.,
Telomere Repeat Sequences
| Species | Repeat Sequence |
|---|---|
| Arabidopsis | TTTAGGG |
| Human | TTAGGG |
| Oxytricha | TTTTGGGG |
| Slime Mold | TAGGG |
| Tetrahymena | TTGGGG |
| Trypanosome | TAGGG |
| Yeast | (TG)1-3TG2-3 |
Notice that the number of TG sequences and the number of cytosines in the yeast sequence varies., Pelo menos para a levedura, foi demonstrado que diferentes estirpes contêm diferentes comprimentos de teleómeros e que o comprimento está sob controle genético.
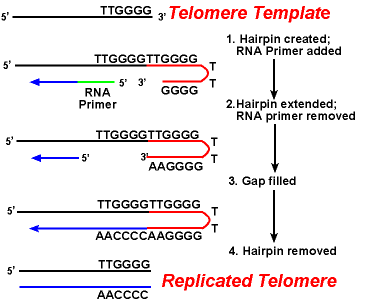
A principal dificuldade com telómeros é a replicação da cadeia retardada.Como a síntese de DNA requer um modelo de RNA (que fornece o grupo 3 ‘ livre) para a replicação de DNA prime, e este modelo é eventualmente degradado, uma pequena região de cadeia única seria deixada no final do cromossomo. Esta região seria susceptível a enzimas que degradam ADN de cadeia simples., O resultado seria que o comprimento do cromossomo seria encurtado após cada divisão. Mas isto não é visto.a acção das enzimas telomerase assegura que as extremidades das cadeias atrasadas são replicadas correctamente. Um sistema bem estudado envolve o organismo protozoário de Tetrahymena. Os telómeros deste organismo terminam na sequência 5′ – TTGGG-3’. A telomerase acrescenta uma série de repetições de 5′-TTGGG-3 ‘ às extremidades da vertente retardada. Um gancho de cabelo ocorre quando pares de base incomuns entre os resíduos de guanina na forma repetida., Em seguida, o iniciador de RNA é removido, e a extremidade 5′ da cadeia retardada pode ser usado para a síntese de DNA. A ligação ocorre entre o fio acabado e o gancho de cabelo. Finalmente, o gancho de cabelo é removido na repetição 5′-TTGGG-3’. Assim, o fim do cromossoma é replicado fielmente. A figura seguinte mostra estes passos.,
A Replicação dos Telômeros
a Análise de Sequências de DNA em Genomas Eucarióticos
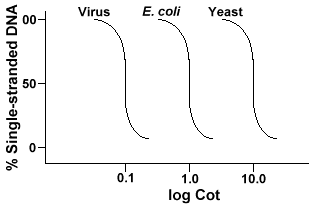
A técnica que é utilizada para determinar a seqüência de complexidade de qualquer genomeinvolves a desnaturação e renaturation de DNA. O ADN é desnaturado por aquecimento que derrete as ligações H e torna o ADN numa cadeia única. Se o ADN for rapidamente arrefecido, o ADN permanece isolado. Mas se o DNAis permitiu arrefecer lentamente, sequências que são complementares irão encontrar um outro par e, eventualmente, base novamente., A taxa a que o DNA reanaleia(outro termo para a renatura) é uma função da espécie a partir da qual o DNAwas isolou. Abaixo está uma curva que é obtida a partir de um genoma simples.
O eixo Y é a percentagem do ADN que permanece em cadeia. É expressa como uma relação entre a concentração de ADN de cadeia simples (C)e a concentração total do ADN inicial (Co). A escala logarítmica de eixo X isa do produto da concentração inicial de ADN (em moles/litro)multiplicada pelo tempo de duração da reacção (em segundos)., The designation for this value is Cot and is called the ” Cot ” value. A curva é chamada de curva “Cot”. Como pode ser visto, a curva é bastante lisa, o que indica que o recozimento ocorre lentamente, mas gradualmente ao longo de um período de tempo. Um valor particular que é útil é o Cot½ , o valor do cot em que metade do ADN voltou a ser selado.passos envolvidos em experiências de desnaturação e Renaturação do ADN. Rasgar o ADN para um tamanho de cerca de 400 bp.
2. Desnaturar o ADN por aquecimento a 100oC.
3. Arrefecer lentamente e colher amostras em intervalos de tempo diferentes.
4., Determinar o % de ADN de cadeia simples em cada momento.
A forma de um “Berço” da curva para uma dada espécie é uma função de dois fatores:
- o tamanho ou a complexidade do genoma; e
- a quantidade de sequências de DNA repetitivas no genoma
Se a gente enredo o “Berço” do que as curvas do genoma de três espécies, tais como bacteriophagelambda, E. coli e levedura vamos ver o que eles têm a mesma forma,mas o Cot½ da levedura vai ser maior, E. coli nextand lambda menor., Fisicamente, quanto maior for o tamanho do genoma, maior será o tempo necessário para que qualquer sequência encontre a sua sequência complementar na solução. Isto porque duas sequências complementares devem encontrar-se umas com as outras antes de poderem fazer pares. Quanto mais complexo for o genoma, ou seja, o número de sequências únicas disponíveis, mais tempo levará para que duas sequências complementares se encontrem e formem pares. Dada a similaridade das concentrações em solução, será necessária uma espécie mais complexa para atingir a Cot½ .,
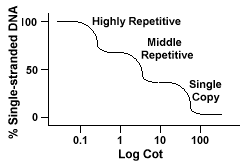
sequências repetitivas de DNA, sequências de DNA que são encontradas mais de uma vez no genoma da espécie, têm efeitos distintos sobre o “Berço” de curvas.Se uma sequência específica for representada duas vezes no genoma, terá duas sequências completamentares a par e, como tal, terá um valor de Cot halfas grande como uma sequência representada apenas uma vez no genoma.
genomas eucarióticos na verdade têm uma grande variedade de sequências que são representadas em diferentes níveis de repetição., Sequências de cópia única são encontradas onceor algumas vezes no genoma. Muitas das sequências que codificam functionalgenes caem nesta classe. O DNA repetitivo médio é encontrado a partir de 10s-1000 vezes no genoma. Exemplos destes incluem rRNA e trnagenos e proteínas de armazenamento em plantas como o milho. DNAcan repetitivo médio varia de 100-300 bp a 5000 bp e pode ser disperso por todo o genoma.As sequências mais abundantes são encontradas no DNAclass altamente repetitivo., Estas sequências são encontradas de 100.000 a 1 milhão de vezes no genomeand pode variar em tamanho de algumas a várias centenas de bases em comprimento. Estas consequências encontram-se em regiões do cromossoma como a heterocromatina,centrómeros e telómeros e tendem a ser organizadas como repetições em tandem. O seguinte é um exemplo de uma sequência repetida em tandemly:
ATTATA ATTATA ATTATA / /ATTATA
genomas que contêm estas diferentes classes de sequências reanalam-se num homem diferente do genoma com apenas sequências de cópia única., Em vez de ter uma única curva suave “Cot”, três curvas distintas podem ser vistas, cada uma representando uma classe de repetição diferente. As primeiras sequências a reanalisar são as sequências altamente repetitivas porque muitas cópias delas existem no genoma, e porque elas têm uma complexidade de sequência baixa. A segunda porção do genoma para reaneal é o DNA repetitivo médio, e a última porção para recaneal é o DNA de cópia única., O diagrama a seguir ilustra o “Berço”da curva para um “típico” eucarióticas genoma
a tabela A seguir apresenta a seqüência de distribuição de espécies selecionadas.
| Espécie | Seqüência de Distribuição |
|---|---|
| Bactérias | 99.,27% Meio Repetitivo 8% Altamente Repetitivas |
| Milho | 30% de Cópia Única 40% Meio Repetitivo 20% Altamente Repetitivas |
| de Trigo | 10% de Cópia Única 83% Meio Repetitivo 4% Altamente Repetitivas |
| Arabidopsis | 55% de Cópia Única 27% Meio Repetitivo 10% Altamente Repetitivas |
a Sequência de Interspersion
Mesmo que os genomas dos organismos superiores conter cópia única, middlerepetitive e altamente repetitivas de seqüências de DNA, estas sequências não são arrangedsimilarly em todas as espécies., O arranjo proeminente é chamado de interpersão de curto prazo. Este arranjo é caracterizado por sequências repetidas de 100 a 200 bp de comprimento intercaladas entre sequências de cópia simples de 1000 a 2000bp de comprimento. Este arranjo é encontrado em animais, fungos e plantas.o segundo tipo de arranjo é a intersecção de longo período. Isto caracteriza-se por 5000 segmentos bp de sequências repetidas intercaladas em regiões de 35 000 bp de ADN de cópia única. Drosophila é uma espécie de planta com flor pertencente à família Fabaceae., Em ambos os casos, as seqüências são geralmente da classe média repetitiva. Discutimos sobre o assunto onde são encontradas sequências altamente repetitivas.
eucariótico cariótipo do cromossoma
enquanto que as bactérias têm apenas um cromossoma único, as espécies eucarióticas têm pelo menos um par de cromossomas. A maioria tem mais de um par. Outro ponto relevante é que os cromossomas eucarióticos só são detectados durante a divisão celular e não durante todas as fases do ciclo celular. Eles estão em sua forma mais condensa durante a metafase quando os cromatídeos irmãos estão ligados.,Este é o estágio primário quando a análise citogenética é realizada.cada espécie é caracterizada por um cariótipo. O cariótipo é a adescrição do número de cromossomas na célula diplóide normal, bem como a sua distribuição de tamanho. Por exemplo, o cromossoma humano tem 23 pares de cromossomas, 22 pares somáticos e um par de cromossomas sexuais. Um aspecto importante da investigação genética está a correlacionar as alterações no cariótipo com as alterações no fenótipo do indivíduo.
Um aspecto importante da genética é correlacionar as alterações no cariótipo com mudanças no fenótipo., Por exemplo, os seres humanos que têm um cromossomo extra 21 têm síndrome de Down. As inserções, supressões e alterações no número de cromossomas podem ser detectadas pelo citogeneticista qualificado, mas é difícil correlacioná-las com fenótipos específicos.
o primeiro parâmetro discriminante ao desenvolver um cariótipo é a dimensão e o número dos cromossomas. Embora isto seja útil, não fornece pormenores suficientes para iniciar o desenvolvimento de uma correlação entre a estrutura e a função (fenótipo)., Para distinguir ainda mais os cromossomas, são tratados com um corante que mancha o ADN de forma reprodutível. Após a formação, algumas das regiões estão levemente manchadas e outras estão amontoadas. Como descrito acima, as regiões levemente manchadas são a calledeuchromatina, e a região manchada escura é chamada deterocromatina. O corante atual de chose é a mancha de Giemsa, e o padrão resultante é chamado de padrão G-banding.,
paradoxo do valor C
além de descrever o genoma de um organismo pelo seu número de cromossomas,também é descrito pela quantidade de ADN numa célula haplóide. Isto é usualmente expresso como a quantidade de DNA por célula haplóide (geralmente expresso como picogramas)ou o número de kilobases por célula haplóide e é chamado de valor C. Uma característica imediata dos organismos eucarióticos realça uma anomalia específica detectada no início da investigação molecular., Embora osorganismos eucarióticos pareçam ter 2 a 10 vezes mais genes do que os procariotas, têm muitas ordens de magnitude mais ADN na célula. Além disso, a quantidade de DNA por genoma está correlacionada não com a presumível complexidade evolutiva de uma espécie.Isto é declarado como o paradoxo do valor C: a quantidade de DNA na haploidcell de um organismo não está relacionada à sua complexidade evolutiva. (Outro ponto importante a ter em mente é que não existe relação entre o número de cromossomas e a presumível complexidade evolutiva de um organismo.,)
C Values of Organisms Used in Genetic Studies
| Species | Kilobases/haploid genome |
|---|---|
| E. coli |
4.5 x 103 |
| Human |
3.0 x 106 |
| Drosophila |
1.7 x 105 |
| Maize |
2.0 x 106 |
| Aribidopsis |
7.,0 x 104 |
Um exemplo dramático da gama de valores de C pode ser visto na planta kingdomwhere Arabidopsis representa a extremidade baixa e lily (1,0 x 10^8 kb/haploidgenome) a extremidade alta da complexidade. Em termos de peso este é 0.07 picogramsper haploid Arabidopsis genoma e 100 picogramas por liligenoma haploid.genoma-o conjunto completo de cromossomas herdados de um único progenitor;o componente completo de ADN de um indivíduo; a definição exclui frequentemente asorganelas