estructura del cromosoma eucariótico
La longitud del ADN en el núcleo es mucho mayor que el tamaño del compartimiento que contiene. Para caber en este compartimiento el ADN tiene que ser condensado de alguna manera. El grado en que se condensa el ADN se expresa como su relación de empaque.
relación de empaquetamiento: la longitud del ADN dividida por la longitud en la que se empaqueta
por ejemplo, el cromosoma humano más corto contiene 4.6 x 107 bp de ADN(aproximadamente 10 veces el tamaño del genoma de E. coli)., Esto equivale a 14.000 µm de ADN extendido. En su estado más condensado durante la mitosis, el cromosoma es de aproximadamente 2 µm de largo. Esto da una relación de empaque de 7000 (14,000/2).
para lograr la relación de empaque general, el ADN no se empaqueta directamente en la estructura final de la cromatina. En cambio, contiene varias jerarquías de organización.El primer nivel de empaquetamiento se logra mediante el enrollado del ADN alrededor de un proteincore para producir una estructura «en forma de grano» llamada nucleosoma. Esto da una relación de embalaje de alrededor de 6., Esta estructura es invariante tanto en la eucromatina como en la heterocromatina de todos los cromosomas. El segundo nivel de empaquetamiento es el enrollado de perlas en una estructura helicoidal llamada fibra de 30 nm que se encuentra tanto en la cromatina de interfase como en los cromosomas mitóticos. Estaestructura aumenta la relación de empaque a aproximadamente 40. El embalaje final se produce cuando la fibra se organiza en bucles, andamios y dominios que dan una relación de embalaje final de aproximadamente 1000 en cromosomas interfásicos y alrededor de 10,000 cromosomas inmitóticos.,
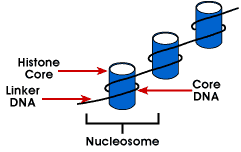
Los cromosomas eucarióticos consisten en un complejo ADN-proteína que se organiza de una manera compacta que permite que la gran cantidad de ADN se almacene en el núcleo de la célula. La designación de subunidad del cromosoma es cromatina.La unidad fundamental de la cromatina es el nucleosoma.,romatina – la unidad de análisis del cromosoma; la cromatina refleja la estructura general del cromosoma, pero no es exclusiva de ningún particulararcromosoma
nucleosoma – estructura de empaquetado más simple del ADN que se encuentra en todos los cromosomas eucarióticos; el ADN está envuelto alrededor de un octámero de pequeñas proteínas básicas llamadas histonas; 146 bp está envuelto alrededor del núcleo y las bases restantes se unen al siguiente nucleosoma; esta estructura causa un superenrollado negativo
el nucleosoma consta de aproximadamente 200 BP envuelto alrededor de un octámero de histonas que contiene dos copias de las proteínas de histonas H2A, H2B, H3 y H4., Estas son conocidas como las histonas del núcleo. Las histonas son proteínas básicas que tienen una afinidadpara el ADN y son las proteínas más abundantes asociadas con el ADN. La secuencia de aminoácidos de estas cuatro histonas se conserva sugiriendo una función similar para la LLA.
la longitud del ADN que se asocia con la unidad de nucleosoma varía entre las especies. Pero independientemente del tamaño, dos componentes de ADN están involucrados. CoreDNA es el ADN que en realidad se asocia con la histona octámero.Este valor es invariante y es de 146 pares de bases., El ADN Central forma dos bucles alrededor del octámero, y esto permite que dos regiones que están separadas por 80 bp se acerquen. Por lo tanto, dos secuencias que están muy separadas pueden interactuar con la misma proteína reguladora para controlar la expresión génica. TheDNA que está entre cada octámero de histona se llama el enlace DNAand puede variar en longitud de 8 a 114 pares de bases. Esta variación es específica, pero la variación en la longitud del ADN enlazador también se ha asociado con la etapa de desarrollo del organismo o regiones específicas del genoma.,
El siguiente nivel de organización de la cromatina es el de la fibra de 30 nm. Esto parece ser una estructura de solenoide con aproximadamente 6 nucleosomas por turno. Esto da una relación de empaque de 40, lo que significa que cada 1 µm a lo largo del eje contiene 40 µm de ADN. La estabilidad de esta estructura requiere la presencia del último miembro de la familia de genes de las histonas, la histona H1., Debido a que los experimentos que eliminan H1 de la cromatina mantienen el nucleosoma, pero no la estructura de 30 nm, se concluyó que H1 es importante para la estabilización de la estructura de 30 nm.
el nivel final de empaquetado se caracteriza por la estructura de 700 nm seenin el cromosoma metafase. La pieza condensada de cromatina tiene una estructura de andamiaje característica que puede detectarse en metafasecromosomas. Esto parece ser el resultado de un extenso bucle de la DNAin el cromosoma.
las últimas definiciones que deben ser presentadas son euchromatin andheterochromatin., Cuando los cromosomas están teñidos con tintes, parecen tener regiones ligeramente teñidas y oscuras. Las regiones ligeramente teñidas son eucromatina y contienen ADN genéticamente activo de una sola copia. Las regiones de tinción oscura son heterocromatina y contienen secuencias repetitivas que son genéticamente inactivas.
centrómeros y telómeros
centrómeros y telómeros son dos características esenciales de todos los cromosomas eucarióticos. Cada uno proporciona una función única que es absolutamente necesaria para la estabilidad del cromosoma., Los centrómeros son necesarios para la segregación del centrómero durante la meiosis y la mitosis, y los teleómeros proporcionan estabilidad terminal al cromosoma y aseguran su supervivencia.
los centrómeros son aquellas regiones condensadas dentro del cromosoma que son responsables de la segregación precisa de los cromosomas replicados durante la mitosis y la meiosis. Cuando los cromosomas se tiñen, por lo general muestran una región teñida de color oscuro que es el centrómero. Durante la mitosis, el centrómero que es compartido por las cromátidas hermanas debe dividirse para que las cromátidas puedan migrar a los polos de la célula., Por otro lado, durante la primera división meiótica, el centrómero de las cromátidas hermanas debe permanecer intacto, mientras que durante la meiosisII deben actuar como lo hacen durante la mitosis. Por lo tanto, el centrómero es un componente importante de la estructura cromosómica y la segregación.
dentro de la región del centrómero, la mayoría de las especies tienen varias ubicaciones donde las fibras del huso se unen, y estos sitios consisten en ADN y proteínas. La ubicación real donde se produce la Unión se llama cinetocoro y está compuesta tanto de ADN como de proteínas. La secuencia de ADN dentro de estas regiones se llama ADN cén., Debido a que el ADN del CEN se puede mover de un cromosoma a otro y aún Así proporcionar al cromosoma la capacidad de segregarse, estas secuencias no deben proporcionar ninguna otra función.
Típicamente el ADN del CEN tiene alrededor de 120 pares de bases y consiste en varios subdominios, CDE-I, CDE-II y CDE-III. Las mutaciones en los dos primeros subdominios no tienen efecto sobre la segregación, pero una mutación puntual en el subdominio CDE-III elimina completamente la capacidad del centrómero para funcionar durante la segregación cromosómica., Por lo tanto, el CDE-III debe participar activamente en la Unión de las fibras del huso al centrómero.
el componente proteico del cinetocoro solo ahora se está caracterizando. Un complejo de tres proteínas llamado Cbf-III se une a las regiones normales de CDE-III, pero no puede unirse a una región de CDE-III con una mutación puntual que impide la segregación mitótica. Además, los mutantes de los genes que codifican las proteínas Cbf-III también eliminan la capacidad de los cromosomas de segregarse durante la mitosis., Análisis adicionales de los componentes de ADN y proteínas del centrómero son necesarios para comprender completamente la mecánica de la segregación cromosómica.
los telómeros son la región del ADN al final del eucariótico lineal que se requiere para la replicación y estabilidad del cromosoma.McClintock reconoció sus características especiales cuando notó que si dos cromosomas se rompían en una célula, el extremo de uno se podía unir al otro y viceversa. Lo que nunca observó fue la Unión del extremo roto al extremo de un cromosoma ininterrumpido., Por lo tanto, los extremos de los cromosomas rotos son pegajosos, mientras que el extremo normal no lo es, lo que sugiere que los extremos de los cromosomas tienen características únicas. Por lo general, pero no siempre, el telomericDNA es heterocromático y contiene secuencias directas repetidas en tándem. La siguiente tabla muestra las secuencias repetidas de varias especies. Estos son a menudo de la forma (T/A)xGy donde x está entre 1 y 4 y y es mayor que 1.,
Telomere Repeat Sequences
| Species | Repeat Sequence |
|---|---|
| Arabidopsis | TTTAGGG |
| Human | TTAGGG |
| Oxytricha | TTTTGGGG |
| Slime Mold | TAGGG |
| Tetrahymena | TTGGGG |
| Trypanosome | TAGGG |
| Yeast | (TG)1-3TG2-3 |
Notice that the number of TG sequences and the number of cytosines in the yeast sequence varies., Al menos para la levadura, se ha demostrado que diferentes cepas contienen diferentes longitudes de teleómeros y que la longitud está bajo control genético.
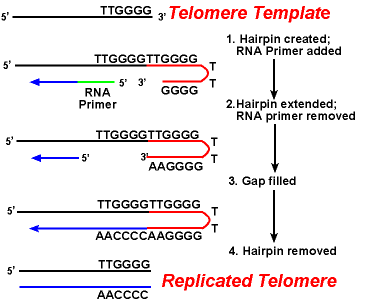
la principal dificultad con los telómeros es la replicación de la cadena rezagada.Debido a que la síntesis de ADN requiere una plantilla de ARN (que proporciona el Grupo 3′-Oh libre) para preparar la replicación del ADN, y esta plantilla se degrada eventualmente, una región corta de una sola cadena se dejaría al final del cromosoma. Esta región sería susceptible a enzimas que degradan el ADN monocatenario., El resultado sería que la longitud del cromosoma se acortaría después de cada división. Pero esto no se ve.
la acción de las enzimas telomerasas asegura que los extremos de las hebras rezagadas se replican correctamente. Un sistema bien estudiado involucra al organismo protozoario Tetrahymena. Los telómeros de este organismo terminan en la secuencia 5 ‘- TTGGGG-3’. La telomerasa añade una serie de repeticiones de 5′-TTGGGG-3 ‘ a los extremos de la hebra rezagada. Una horquilla ocurre cuando pares de bases inusuales entre residuos de guanina en la forma repetida., A continuación, se elimina la imprimación de ARN, y el extremo 5′ de la hebra rezagada se puede utilizar para la síntesis de ADN. La ligadura ocurre entre la hebra retrasada terminada y la horquilla. Finalmente, la horquilla se retira en la repetición de 5′-TTGGGG-3’. Así, el extremo del cromosoma se replica fielmente. La siguiente figura muestra estos pasos.,
la replicación de telómeros
análisis de secuencias de ADN en genomas eucarióticos
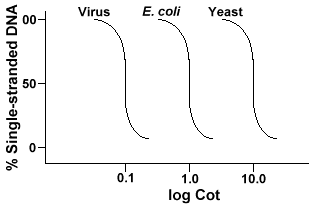
la técnica que se utiliza para determinar la complejidad de secuencia de cualquier genoma implica la desnaturalización y renaturalización del ADN. El ADN es desnaturalizado por calentamiento que funde los enlaces H y hace que el ADN sea monocatenario. Si el ADN se enfría rápidamente, el ADN permanece monocatenario. Pero si el DNAis permitió enfriar lentamente, las secuencias que son complementarias se encontrarán entre sí y eventualmente par de bases nuevamente., La velocidad a la que el ADN se reanuda(otro término para renatura) es una función de la especie de la que el DNAwas aislado. A continuación se muestra una curva que se obtiene de un genoma simple.
el eje Y es el porcentaje del ADN que permanece monocatenario. Esto se expresa como una relación entre la concentración de ADN monocatenario (C)y la concentración total del ADN inicial (Co). El eje X es una escala logarítmica del producto de la concentración inicial de ADN (en moles/litro)multiplicada por el tiempo que la reacción continuó (en segundos)., La designaciónpara este valor es Cot y se llama el valor «Cot». La curva en sí misma se llama curva «Cot». Como se puede ver, la curva es bastante suave, lo que indica que el recocido se produce lentamente, pero gradualmente durante un período de tiempo. Un valor en particular que es útil es Cot½, el valor de la olla donde la mitad del ADN se ha recocido.
pasos involucrados en los experimentos de desnaturalización y renaturalización del ADN
1. Corte el ADN a un tamaño de unos 400 bp.
2. Desnaturalizar el ADN calentándolo a 100oC.3. Enfriar lentamente y tomar muestras a diferentes intervalos de tiempo.
4., Determinar el % de ADN monocatenario en cada punto de tiempo.
la forma de una curva «Cot» para una especie dada es una función de dos factores:
- El tamaño o complejidad del genoma; y
- La cantidad de ADN repetitivo dentro del genoma
si trazamos las curvas «Cot» del genoma de tres especies como bacteriophagelambda, E. coli y levadura veremos que tienen la misma forma,pero la Cot½ de la levadura será más grande, E. coli la siguiente y lambda más pequeña., Físicamente, cuanto mayor sea el tamaño del genoma, más tardará una secuencia en encontrar su secuencia complementaria en la solución. Esto se debe a que dos secuencias complementarias deben encontrarse antes de que puedan emparejarse. Cuanto más complejo sea el genoma, es decir, las secuencias más únicas que están disponibles, más tiempo tomará para que dos secuencias complementarias se encuentren y se emparejen. Dadas concentraciones similares en solución, tardará una especie más compleja en llegar a Cot½ .,
Las secuencias de ADN repetidas, secuencias de ADN que se encuentran más de una vez en el genoma de la especie, tienen efectos distintivos en las curvas «Cot».Si una secuencia específica se representa dos veces en el genoma, tendrá dos secuencias complementarias para emparejarse y, como tal, tendrá un valor de Cot medio grande como una secuencia representada solo una vez en el genoma.
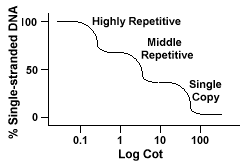
los genomas eucarióticos en realidad tienen una amplia gama de secuencias que se representan en diferentes niveles de repetición., Secuencias de una sola copia se encuentran una vez o un par de veces en el genoma. Muchas de las secuencias que codifican funcionalgenes caen en esta clase. El ADN medio repetitivo se encuentra entre 10 y 1000 veces en el genoma. Ejemplos de estos incluyen rRNA y trnagenos y proteínas de almacenamiento en plantas como el maíz. El DNAcan medio repetitivo varía de 100-300 bp a 5000 bp y puede dispersarse por todo el genoma.Las secuencias más abundantes se encuentran en la altamente repetitiva Dnaclase., Estas secuencias se encuentran de 100.000 a 1 millón de veces en el genoma y pueden variar en tamaño de unos pocos a varios cientos de bases en longitud. Estas consecuencias se encuentran en regiones del cromosoma como heterocromatina, centrómeros y telómeros y tienden a estar dispuestos como repeticiones en tándem. El siguiente es un ejemplo de una secuencia repetida tándem:
ATTATA ATTATA ATTATA / /ATTATA
genomas que contienen estas diferentes clases de secuencias reaneal en un escáner diferente que los genomas con secuencias de copia única., En lugar de tener una única curva «Cot» Lisa, se pueden ver tres curvas distintas, cada una representando una clase de repetición diferente. Las primeras secuencias que se recobran son las secuencias altamente repetitivas porque existen tantas copias de ellas en el genoma,y porque tienen una baja complejidad secuencial. La segunda porción del genoma a reconducir es el ADN repetitivo medio, y la porción final a recodar es el ADN de copia única., El siguiente diagrama representa la curva » Cot «para un genoma eucariótico «típico»
la siguiente tabla da la distribución de la secuencia para especies seleccionadas.
| Species | Sequence Distribution |
|---|---|
| Bacteria | 99.,27% repetitivo medio 8% altamente repetitivo |
| maíz | 30% copia única 40% repetitivo medio 20% altamente repetitivo |
| trigo | 10% copia única 83% repetitivo medio 4% altamente repetitivo |
| Arabidopsis | 55% copia única 27% repetitivo medio 10% altamente repetitivo |
intercesión de secuencias
aunque los genomas de los organismos superiores contienen secuencias de ADN de copia única, medio repetitivo y altamente repetitivo, estas secuencias no están dispuestas de manera similar en Todas las especies., La disposición prominente se llama período cortointerspersión. Esta disposición se caracteriza por secuencias100-200 bp repetidos en longitud intercalados entre secuencias de copia única que son 1000-2000bp en longitud. Esta disposición se encuentra en animales, hongos y plantas.
el segundo tipo de arreglo es la intercalación de largo plazo. Esto se caracteriza por tramos de 5000 PB de secuencias repetidas intercaladas en regiones de 35.000 PB de ADN de copia única. Drosophila es un ejemplo de una especie con esta disposición secuencial poco común., En ambos casos, las secuencias tratadas son generalmente de la clase media repetitiva. Discutimos sobre dónde se encuentran secuencias altamente repetitivas.
cariotipo del cromosoma eucariótico
mientras que las bacterias solo tienen un solo cromosoma, las especies eucariotas tienen al menos un par de cromosomas. La mayoría tiene más de un par. Otro punto relevante es que los cromosomas eucarióticos se detectan solo se producen durante la división celular y no durante todas las etapas del ciclo celular. Están en su forma más condensada durante la metafase cuando las cromátidas hermanas están unidas.,Este es el estadio primario cuando se realiza el análisis citogenético.
Cada especie se caracteriza por un cariotipo. El cariotipo es una descripción del número de cromosomas en la célula diploide normal, así como su distribución de tamaño. Por ejemplo, el cromosoma humano tiene 23 pares de cromosomas, 22 pares somáticos y un par de cromosomas sexuales. Un aspecto importante de la investigación genética es la correlación de los cambios en el cariotipo con los cambios en el fenotipo del individuo.
un aspecto importante de la genética es la correlación de los cambios en el cariotipo con los cambios en el fenotipo., Por ejemplo, los seres humanos que tienen un cromosoma 21 adicional tienen síndrome de Down. El citogenetista experto puede detectar inserciones, deleciones y cambios en el número de cromosomas, pero es difícil correlacionarlos con fenotipos específicos.
el primer parámetro que discrimina cuando se desarrolla un cariotipo es el tamaño y número de los cromosomas. Aunque esto es útil, no proporciona suficiente detalle para comenzar el desarrollo de una correlación entre estructura y función (fenotipo)., Para distinguir aún más entre los cromosomas, se tratan con un tinte que tiñe el ADN de manera reproducible. Después de la tinción, algunas de las regiones están ligeramente manchadas y otras están fuertemente manchadas. Como se describió anteriormente, las regiones ligeramente teñidas se llaman euchromatina, y la región teñida oscura se llama heterocromatina. El tinte actual de chose es la tinción de Giemsa,y el patrón resultante se llama el patrón de bandas G.,
paradoja del valor C
Además de describir el genoma de un organismo por su número de cromosomas, también se describe por la cantidad de ADN en una célula haploide. Esto se expresa generalmente como la cantidad de ADN por célula haploide (generalmente expresada como picogramos) o el número de kilobases por célula haploide y se llama el valor C. Una característica inmediata de los organismos eucariotas destaca una anomalía específica que se detectó al principio de la investigación molecular., A pesar de que los organismos eucariotas parecen tener 2-10 veces más genes que los procariotas, tienen muchos ordenesde magnitud más ADN en la célula. Además, la cantidad de ADN por genoma no se correlaciona con la supuesta complejidad evolutiva de una especie.Esto se afirma como la paradoja del valor C: la cantidad de ADN en la haploidcell de un organismo no está relacionada con su complejidad evolutiva. (Otro punto importante a tener en cuenta es que no existe relación entre el número de cromosomas y la supuesta complejidad evolutiva de un organismo.,)
C Values of Organisms Used in Genetic Studies
| Species | Kilobases/haploid genome |
|---|---|
| E. coli |
4.5 x 103 |
| Human |
3.0 x 106 |
| Drosophila |
1.7 x 105 |
| Maize |
2.0 x 106 |
| Aribidopsis |
7.,0 x 104 |
Un ejemplo dramático del rango de valores de C se puede ver en el rey de la planta donde Arabidopsis representa el extremo inferior y lily (1.0 x 10^8 kb/haploidgenome) el extremo superior de la complejidad. En términos de peso esto es 0.07 picogramsper genoma haploide de Arabidopsis y 100 picogramos por liligenoma haploide.
genoma-el conjunto completo de cromosomas heredados de un padre soltero;el componente de ADN completo de un individuo; la definición a menudo excluye organuelles